1. Comprensión inicial
Nesta fase, necesitamos comprender algúns conceptos e terminoloxía, para evitar cometer erros diante dos nosos maiores, como:
P: Cal é a diferenza entre RT-PCR, qPCR, PCR en tempo real e RT-PCR en tempo real?
Resposta: RT-PCR é PCR de transcrición inversa(PCR de transcrición inversa, RT-PCR), que é unha variante moi utilizada da reacción en cadea da polimerase (PCR).Na RT-PCR, unha cadea de ARN transcríbese en sentido inverso a ADN complementario, que despois se usa como molde para a amplificación de ADN mediante PCR.
PCR en tempo real e qPCR(Quantitative Rea-ltime-PCR) son o mesmo, ambos son PCR cuantitativos en tempo real, o que significa que cada ciclo de PCR ten rexistros de datos en tempo real, polo que o número de modelos iniciais pode axustarse á análise precisa.
Aínda que tanto a PCR en tempo real (PCR cuantitativa fluorescente en tempo real) como a PCR de transcrición inversa (PCR de transcrición inversa) parecen abreviarse como RT-PCR, a convención internacional é: RT-PCR refírese específicamente á transcrición inversa.PCR, a PCR en tempo real abreviase xeralmente como qPCR (PCR cuantitativa en tempo real).
E RT-PCR en tempo real (RT-qPCR), é a PCR de transcrición inversa combinada coa tecnoloxía cuantitativa fluorescente: primeiro obtén ADNc (RT) da transcrición inversa do ARN e despois usa a PCR en tempo real para a análise cuantitativa (qPCR).A maioría dos laboratorios fan RT-qPCR, é dicir, investigacións sobre a regulación á baixa da expresión do ARN, polo que a qPCR da que todo o mundo fala no laboratorio refírese en realidade á RT-qPCR, pero non esquezas que aínda hai moitas probas de ADN en aplicacións clínicas.Análise cuantitativa, como a detección do VHB do virus da hepatite B.
Pregunta: Despois de ler moita PCR cuantitativa fluorescente, por que debería controlarse o fragmento amplificado dentro do intervalo de 80-300 pb?
Resposta: A lonxitude de cada secuencia de xenes é diferente, algunhas son varios kb, outras son centos de pb, pero só precisamos que a lonxitude do produto sexa de 80-300 pb cando se deseña cebadores, demasiado curtos ou demasiado longos non son axeitados para a detección cuantitativa por PCR fluorescente.O fragmento do produto é demasiado curto para poder distinguilo do cebador-dímero.A lonxitude do cebador-dímero é de aproximadamente 30-40 pb, e é difícil distinguir se é un cebador-dímero ou un produto se é inferior a 80 pb.Se o fragmento do produto é demasiado longo, superando os 300 pb, provocará facilmente unha baixa eficiencia de amplificación e non poderá detectar eficazmente a cantidade do xene.
Por exemplo, cando contas cantas persoas hai nunha aula, só tes que contar cantas bocas hai.O mesmo ocorre cando detectas xenes, só tes que detectar unha determinada secuencia dun xene para representar Toda a secuencia servirá.Se queres contar persoas, hai que contar tanto a boca como o nariz, as orellas e as lentes, e é fácil cometer erros.
Para ampliar, na investigación biolóxica, hai moitos casos de investigación dun punto a outro, porque a secuencia xenética de calquera especie é moi longa, é innecesario e imposible medir todos os fragmentos, como a secuenciación bacteriana 16S, que consiste en realizar a secuencia conservadora de Ensaios de bacterias para inferir o número dunha determinada poboación de bacterias.
P: Cal é a lonxitude óptima para o deseño do cebador de qPCR?
Resposta: En xeral, a lonxitude do cebador é de aproximadamente 20-24 pb, o que é mellor.Por suposto, debemos prestar atención ao valor TM da imprimación á hora de deseñar a imprimación, porque esta está relacionada coa temperatura óptima de recocido.Despois de moitos experimentos, comprobouse que 60 °C é un mellor valor de TM.Se a temperatura de recocido é demasiado baixa, facilmente levará a unha amplificación inespecífica.Se a temperatura de recocido é demasiado alta, a eficiencia de amplificación será relativamente baixa, o pico da curva de amplificación comezará máis tarde e o valor CT atrasarase.
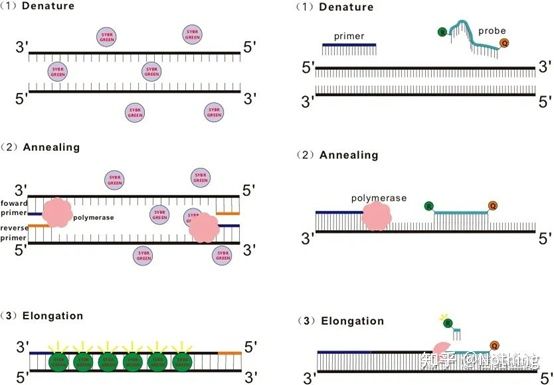
P: Como é diferente o método de tintura do método de sonda?
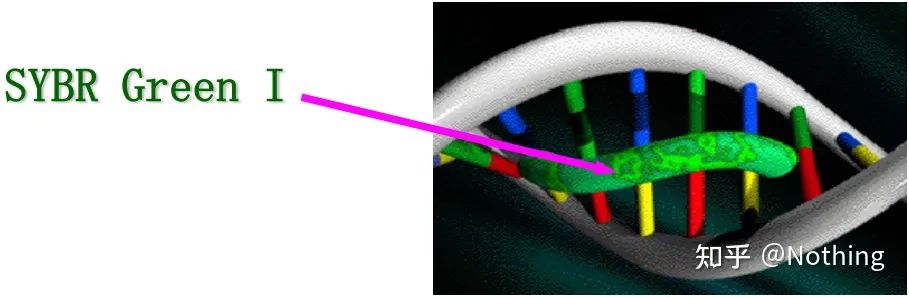
Resposta: Método de tinturaAlgúns colorantes fluorescentes, como SYBR Green Ⅰ, PicoGreen, BEBO, etc., non emiten luz por si mesmos, pero emitirán fluorescencia despois de unirse ao suco menor do ADN de dobre cadea.Polo tanto, ao comezo da reacción de PCR, a máquina non pode detectar o sinal fluorescente.Cando a reacción chega á fase de recocido-extensión, ábrese a dobre cadea e sintetízase unha nova cadea baixo a acción da ADN polimerase, e a molécula fluorescente únese ao suco menor do dsDNA.A medida que aumenta o número de ciclos de PCR, combínanse máis e máis colorantes con ADN de dobre cadea e o sinal fluorescente tamén se mellora continuamente.O método de tintura úsase principalmente na investigación científica.
PD: Teña coidado ao facer o experimento, o colorante ten que combinarse co ADN humano, teña coidado de convertelo nunha persoa fluorescente.
Método de tintura (esquerda) Método de sonda (dereita)
PD: Teña coidado ao facer o experimento, o colorante ten que combinarse co ADN humano, teña coidado de convertelo nunha persoa fluorescente.
SYBR Green Ⅰ únese ao suco menor do ADN
Método da sondaA sonda Taqman é a sonda de hidrólise máis utilizada.Hai un grupo fluorescente no extremo 5′ da sonda, xeralmente FAM, e a propia sonda é unha secuencia complementaria ao xene diana.Hai un grupo de extinción fluorescente no extremo 3′.Segundo o principio de transferencia de enerxía de resonancia de fluorescencia (Förster resonance energy transfer, FRET), cando o grupo fluorescente informador (molécula fluorescente doadora) e o grupo fluorescente de extinción (molécula fluorescente aceptora) son excitados. Cando os espectros se solapan e a distancia é moi próxima (7-10 nm), a excitación da molécula de fluorescencia, mentres que a molécula de fluorescencia doador acepta a molécula de fluorescencia. nce está debilitado.Polo tanto, ao comezo da reacción de PCR, cando a sonda está libre e intacta no sistema, o grupo fluorescente informador non emitirá fluorescencia.Ao recocer, o cebador e a sonda únense ao molde.Durante a etapa de extensión, a polimerase sintetiza continuamente novas cadeas.A ADN polimerase ten actividade exonuclease 5′-3′.Ao chegar á sonda, a ADN polimerase hidrolizará a sonda do molde, separará o grupo fluorescente informador do grupo fluorescente extintor e liberará o sinal fluorescente.Dado que existe unha relación un a un entre a sonda e a plantilla, o método da sonda é superior ao método da tintura en canto á precisión e sensibilidade da proba.O método de sonda emprégase principalmente no diagnóstico.
P: Que é a cuantificación absoluta?Que é a cuantificación relativa?
Resposta: A cuantificación absoluta refírese ao cálculo do número de copia inicial da mostra que se vai probar mediante qPCR, como cantos virus do VHB hai en 1 ml de sangue.O resultado obtido pola cuantificación relativa é o cambio na cantidade do xene diana nunha mostra específica en relación con outra mostra de referencia, e a expresión xénica está regulada á alza ou á baixa.
P: A cantidade de extracción de ARN, a eficiencia da transcrición inversa e a eficiencia da amplificación afectarán os resultados experimentais?
P: Afectarán os resultados experimentais o almacenamento de mostras, os reactivos de extracción, os reactivos de transcrición inversa e os consumibles de transmisión de luz?
P: Que método pode corrixir os datos experimentais?
Respecto a estes problemas, describirémolos en detalle nas seccións avanzadas e avanzadas a continuación.
2. Coñecementos avanzados
No que respecta á PCR cuantitativa fluorescente en tempo real, hai que recoñecer a realidade de que cada ano se publican miles de traballos de investigación científica, entre os que a tecnoloxía de PCR cuantitativa fluorescente non é pouco.
Se non existe un estándar común para medir o experimento de PCR cuantitativa fluorescente, os resultados poden variar moito.Para o mesmo xene da mesma especie, co mesmo método de procesamento, os resultados da detección tamén variarán moito, e será difícil para os que chegan tarde repetir os mesmos resultados.Ti Ninguén sabe cal é correcto e cal está mal.
Significa isto que a PCR cuantitativa fluorescente é unha tecnoloxía fraudulenta ou unha tecnoloxía pouco fiable?Non, é porque a PCR cuantitativa fluorescente é máis sensible e máis precisa, e unha pequena operación incorrecta producirá resultados completamente opostos.Unha pequena perda está a mil millas de distancia.O autor do artigo pode ser torturado repetidamente polos revisores.Ao mesmo tempo, os revisores da revista tamén son difíciles de escoller entre diferentes resultados experimentais.
En definitiva, sinalando a falta de consenso nos experimentos de PCR en tempo real.Para este fin, científicos seniores da industria comezaron a formular estándares,esixindo aos colaboradores que proporcionen algúns detalles experimentais e de procesamento de datos necesarios (incluídos os datos necesarios) no artigo para cumprir estes estándares.
Os revisores poden xulgar a calidade do experimento lendo estes detalles;os futuros lectores tamén poden usalo para repetir o experimento ou mellorar o experimento.Entón os resultados experimentais obtidos deste xeito están cheos de información, de alta calidade e utilizables.
MIBBI (Información mínima para investigacións biolóxicas e biomédicas)http://www.mibbi.org) xurdiu.MIBBI é un proxecto que proporciona estándares para experimentos.Está publicado na natureza.Este proxecto está dirixido a varios experimentos biolóxicos, entre eles a bioloxía celular, Microarray, qPCR que imos comentar agora, etc., e prevé cada tipo de experimento á hora de enviar manuscritos.Esa información debe proporcionarse en todo momento.
No proxecto MIBBI, hai dous artigos relacionados coa PCR cuantitativa fluorescente, a saber:
·RDML (Real-Time PCR Data Markup Language): unha linguaxe estruturada e unha guía de informes para datos de PCR cuantitativos en tempo real;
·MIQE (Minimum Information for Publication of Quantitative Real-Time PCR Experiments) – información mínima para publicar artigos sobre experimentos de PCR cuantitativos en tempo real.
En primeiro lugar, imos falar de RDML, a especificación terminolóxica.
Se non hai unha definición estándar para todo, é imposible continuar coa discusión, polo que a explicación dos termos é tan importante no exame.
A terminoloxía utilizada no experimento de PCR cuantitativa fluorescente inclúe o seguinte contido.QIAGEN fixo o mellor resumo para nós.Os seguintes están todos secosmercadorías .
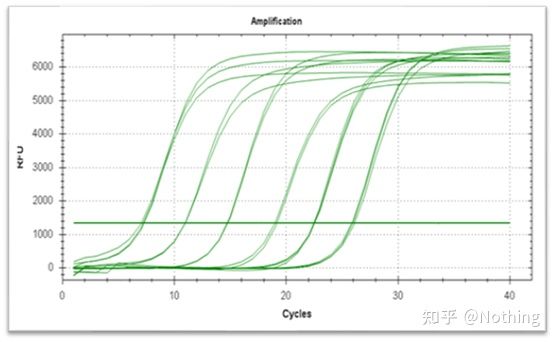
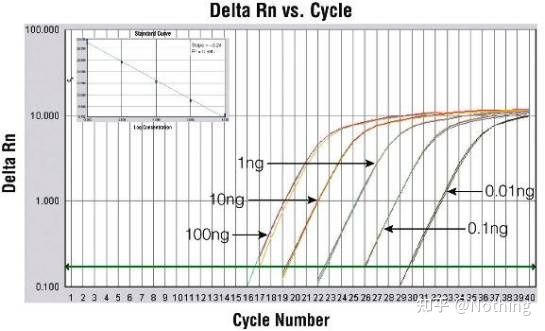
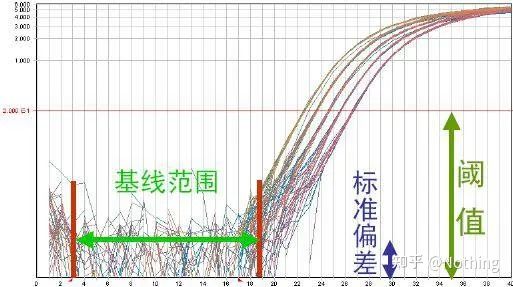
Curva de amplificación
A curva de amplificación refírese á curva realizada durante o proceso de PCR, co número de ciclo como abscisa e a intensidade de fluorescencia en tempo real durante a reacción como ordenada.
Unha excelente curva de amplificación debe ter as seguintes características: a liña de base é plana ou lixeiramente diminuída e non hai unha tendencia ascendente obvia;o punto de inflexión da curva é claro e a pendente da fase exponencial é proporcional á eficiencia de amplificación.Canto maior sexa a pendente, maior será a eficiencia de amplificación;a curva de amplificación global O paralelismo é bo, o que indica que a eficiencia de amplificación de cada tubo é similar;a fase exponencial da curva de amplificación de mostras de baixa concentración é obvia.
Liña de base (línea de base)
A liña de base é o nivel de ruído do ciclo inicial, normalmente medido entre o 3o e o 15o ciclo, porque o aumento do valor de fluorescencia provocado polo produto de amplificación non se pode detectar durante este período.O número de ciclos utilizados para calcular a liña de base pode variar e pode ter que reducirse se se usan grandes cantidades de molde ou se o nivel de expresión do xene diana é alto.
Establecer a liña de base require ver os datos de fluorescencia da curva de amplificación da linealidade .A liña base establécese de xeito que o crecemento da curva de amplificación comece cun número de ciclo maior que o número superior do ciclo base.As liñas de base deben establecerse individualmente para cada secuencia obxectivo.Os valores medios de fluorescencia detectados nos primeiros ciclos hai que restar dos valores de fluorescencia obtidos nos produtos amplificados.As últimas versións de varios programas de PCR en tempo real permiten a optimización automática da configuración de referencia para mostras individuais.
Durante os primeiros ciclos da reacción de amplificación da PCR, o sinal de fluorescencia non cambia moito.Aproximarse a unha liña recta chámase liña de base, pero se observamos detidamente os primeiros ciclos, vemos que dentro da liña de base está o que está a suceder na imaxe de abaixo.
Fondo Refírese ao fondo
o valor de fluorescencia inespecífico na reacción.Por exemplo: extinción de fluorescencia ineficiente;ou un gran número de modelos de ADN de dobre cadea debido ao uso de SYBR Green.Os compoñentes de fondo do sinal son eliminados matematicamente polo algoritmo de software de PCR en tempo real.
Sinal do reporteiro
O sinal informador refírese ao sinal fluorescente xerado por SYBR Green ou sondas específicas de secuencia marcadas con fluorescencia durante a PCR en tempo real.
Sinal de informador normalizado (RN)
RN refírese á intensidade de fluorescencia do colorante indicador dividida pola intensidade de fluorescencia do colorante de referencia pasivo medida en cada ciclo.
Tintura de referencia pasiva
Nalgunhas PCR en tempo real,o colorante fluorescente ROX úsase como referencia interna para normalizar o sinal fluorescente.Corrixe as variacións debidas ao pipeteo inexacto, á posición do pozo e ás flutuacións de fluorescencia pozo por pozo.
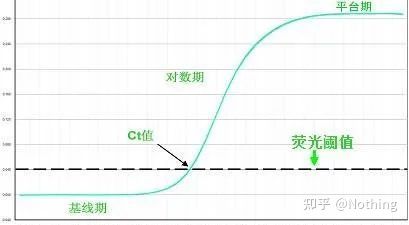
Limiar de fluorescencia (umbral)
axustouse por riba do valor de fondo e significativamente por debaixo do valor da meseta da curva de amplificación.Debe situarse na rexión lineal da curva de amplificación, que representa o intervalo logarítmico lineal de detección por PCR.Os limiares deben establecerse na vista da curva de amplificación logarítmica para que a fase logarítmica lineal da PCR sexa facilmente identificable.Se hai varios xenes diana na PCR en tempo real, debe establecerse o limiar para cada diana.Xeralmente, o sinal de fluorescencia dos primeiros 15 ciclos de reacción de PCR úsase como sinal de fondo de fluorescencia, e o limiar de fluorescencia é 10 veces a desviación estándar do sinal de fluorescencia dos primeiros 3 a 15 ciclos de PCR, e o limiar de fluorescencia establécese na fase de amplificación exponencial da PCR.En xeral, cada instrumento ten establecido o seu limiar de fluorescencia antes do seu uso.
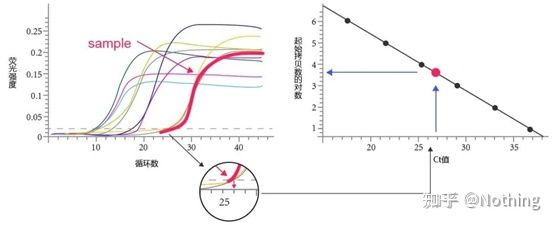
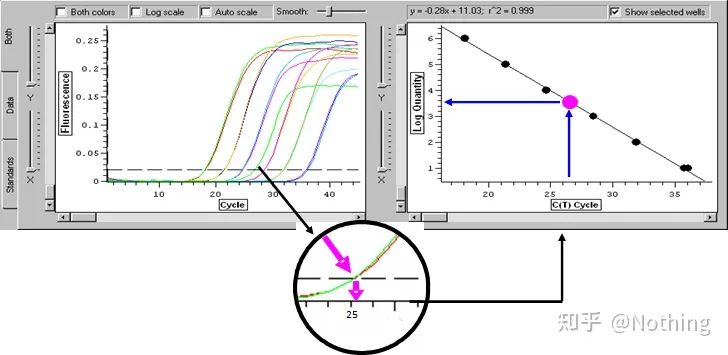
Limiar de ciclo (CT) ou punto de cruce (CP)
O ciclo no que a curva de amplificación cruza o limiar (é dicir, o punto no que a detección de fluorescencia aumenta significativamente).CT pode ser unha fracción e pódese calcular a cantidade de modelo inicial.O valor CT representa o número de ciclos experimentados cando o sinal fluorescente en cada tubo de reacción de PCR alcanza o limiar establecido.Existe unha relación lineal entre o valor CT de cada modelo e o logaritmo do número de copia inicial do modelo, omaior será o número de copia inicial, menor será o valor de CT e viceversa.Pódese facer unha curva estándar usando un estándar cun número de copia inicial coñecido, onde a abscisa representa o valor CT e a ordenada representa o logaritmo do número de copia inicial.Polo tanto, sempre que se obteña o valor CT da mostra descoñecida, o número de copia inicial da mostra pódese calcular a partir da curva estándar.
Valor ΔCT
O valor ΔCT describea diferenza entre o xene obxectivo e o valor CT do xene de referencia endóxeno correspondente, como un xene doméstico, e úsase para normalizar a cantidade de modelo utilizado:
⇒ΔCT = CT (xene obxectivo) – CT (xene de referencia endóxeno)
Valor ΔΔCT
O valor ΔΔCT describe a diferenza entre o valor medio ΔΔCT dunha mostra de interese (por exemplo, células estimuladas) e o valor medio ΔΔCT dunha mostra de referencia (por exemplo, células non estimuladas).A mostra de referencia tamén se denomina mostra de calibración e todas as demais mostras normalízanse para a súa cuantificación relativa:
⇒ΔΔCT = ΔCT medio (mostra de interese) – ΔCT medio (mostra de referencia)
Xenes de referencia endóxenos (xenes de referencia endóxenos)
Os niveis de expresión dos xenes de referencia endóxenos, como os xenes domésticos (xenes domésticos), non difiren entre as mostras.A comparación dos valores de CT do xene de referencia co xene diana permite que o nivel de expresión do xene diana se normalice coa cantidade de ARN ou ADNc de entrada (consulte a sección sobre os valores de ΔCT anterior).
Xenes de referencia internos correctos paraa posible degradación do ARN ou a presenza de inhibidores enzimáticos nas mostras de ARN, así como as variacións no contido de ARN, a eficiencia da transcrición inversa, a recuperación de ácidos nucleicos e o manexo das mostras.Para seleccionar o xene(s) de referencia óptimo(s), modificamos o algoritmo para permitir a súa selección da referencia óptima dependendo da configuración experimental.
Control interno
Unha secuencia de control que se amplifica na mesma reacción que a secuencia diana e que se sonda cunha sonda diferente (é dicir, realizando unha PCR dúplex).Os controis internos úsanse a miúdo para descartar amplificacións fallidas, como cando non se detecta a secuencia diana.
Mostra de calibración
Unha mostra de referencia (por exemplo, ARN purificado dunha liña celular ou tecido) utilizada na cuantificación relativa para comparar todas as demais mostras para determinar o nivel de expresión relativo dun xene.A mostra de calibración pode ser calquera mostra, pero adoita ser un control (por exemplo, unha mostra non tratada ou unha mostra do tempo cero do experimento).
Controis positivos
utilizar reaccións de control concantidades coñecidas de modelo.Os controis positivos úsanse a miúdo para comprobar que un conxunto de cebadores ou un conxunto de cebadores-sonda funcionan correctamente e que a reacción está configurada correctamente.
Sen control de modelos (NTC)
Reacción de control que contén todos os compoñentes necesarios da reacción de amplificación, excepto a plantilla, que adoita ser substituída por auga.O uso de NTC pode atopar a contaminación causada pola contaminación dos reactivos ou por ADN estraño, garantindo así a autenticidade e fiabilidade dos datos de detección.A amplificación do control NTC indica contaminación.
Sen control RT (NRT)
O proceso de extracción de ARN pode conter ADN xenómico residual, que é extremadamente prexudicial e é o culpable de afectar á calidade dos datos e o inimigo natural da qPCR, polo que ao deseñar experimentos, debe deseñarse só para amplificar a detección de ARN.Hai dúas formas, unha é deseñar cebadores entre intróns, a outra é eliminar completamente o ADN, cal é mellor, que se comentará máis adiante.O control NTR é un espello máxico para detectar a contaminación do ADN.Se hai amplificación, significa que hai contaminación.
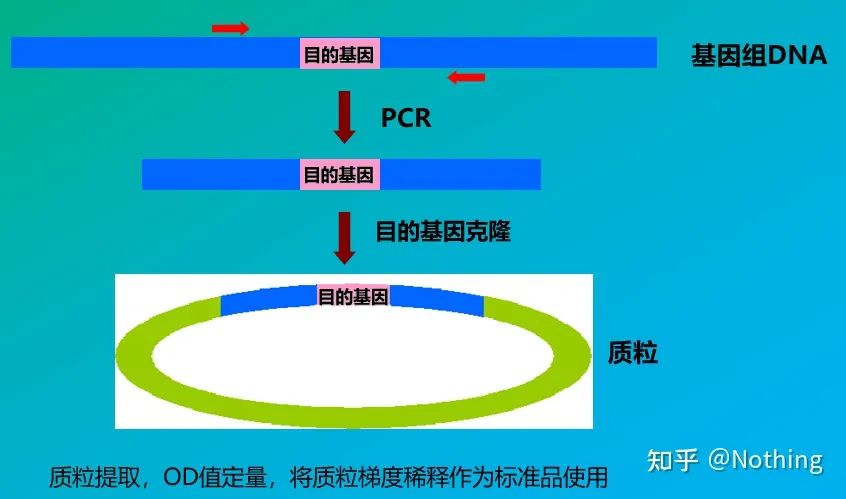
Estándares
Os patróns son mostras de concentración coñecida ou número de copias que se usan para construír unha curva estándar.Para garantir a estabilidade do estándar, o fragmento xene adoita ser clonado no plásmido e utilízase como estándar.
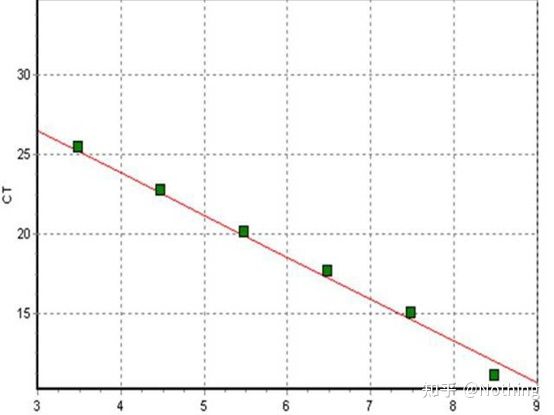
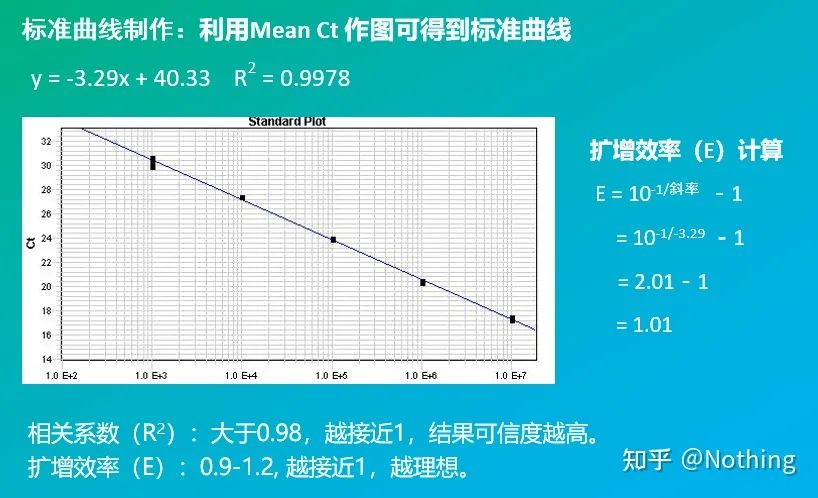
A curva estándar
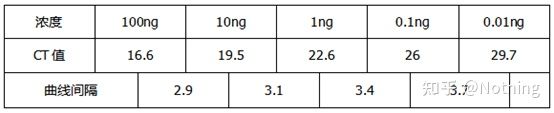
adoita dilúese en polo menos 5 gradientes de concentración co produto estándar segundo a relación de duplicación, e 5 puntos son debuxados nas coordenadas do valor CT e número de copia, e os puntos están conectados para formar unha liña para xerar unha curva estándar.Para cada curva estándar, hai que comprobar a súa validez.O valor da pendente sitúase entre –3,3 e –3,8, e cada concentración realízase por triplicado.Os puntos que sexan significativamente diferentes doutros puntos deben ser descartados.O valor CT da mostra que se vai probar lévase á curva estándar e pódese calcular o nivel de expresión da mostra que se vai probar.
O valor CT da mostra que se vai probar lévase á curva estándar e pódese calcular o número de copia inicial da mostra que se vai probar.
Eficiencia e Pendente
A pendente da curva estándar representa a eficiencia da PCR en tempo real.
·Unha pendente de -3,322 indica que a eficiencia da amplificación da PCR é 1, ou 100% eficiente, e a cantidade de produto da PCR duplícase en cada ciclo.
·Unha pendente inferior a –3,322 (por exemplo, –3,8) indica unha eficiencia da PCR
·Unha pendente superior a –3,322 (por exemplo, –3,0) indica que a eficiencia da PCR parece ser superior ao 100%, o que é curioso, como podería un ciclo de PCR xerar máis do dobre do produto amplificado?Esta situación ocorre na fase non lineal da reacción de PCR, é dicir, hai unha gran cantidade de amplificación inespecífica.
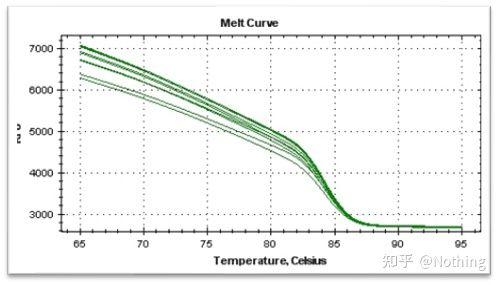
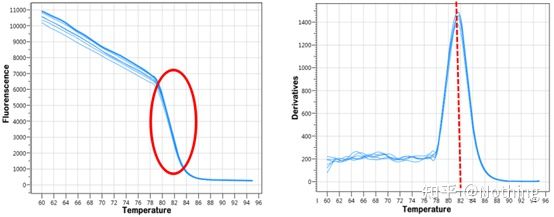
curva de fusión
Despois de completar a amplificación qPCR, o produto da PCR quéntase.A medida que aumenta a temperatura, o produto de amplificación de dobre cadea fúndese gradualmente, o que resulta nunha diminución da intensidade da fluorescencia.Cando se alcanza unha determinada temperatura (Tm), un gran número de produtos fundiranse.A fluorescencia cae bruscamente.Diferentes produtos de PCR teñen diferentes valores de Tm e diferentes temperaturas de fusión, polo que se pode identificar a especificidade da PCR.
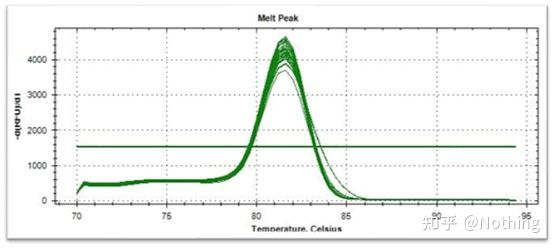
Curva de fusión (curva derivada)
A curva de fusión derívase para formar un mapa de picos, que pode mostrar de forma máis intuitiva a situación dos fragmentos de produtos da PCR.Dado que a temperatura de fusión é o valor de Tm do fragmento de ADN, pódense xulgar algúns parámetros que afectan o valor de Tm do fragmento de ADN, como o tamaño do fragmento, o contido de GC, etc. En xeral, segundo os nosos principios de deseño de cebadores,a lonxitude do produto amplificado está no rango de 80-300 pb, polo que a temperatura de fusión debe estar entre 80 °C e 90 °C.
Interpretación da curva de fusión: Se o único pico principal aparece entre 80°C-90°C, significa que a PCR cuantitativa fluorescente é perfecta;se o pico principal aparece entre os 80°C-90°C e os picos varios aparecen por debaixo dos 80°C, considérase basicamente o dímero de cebador.Podes tentar aumentar a temperatura de recocido para solucionalo;se o pico principal aparece entre 80°C e 90°C, e o pico misceláneo aparece de novo cando a temperatura aumenta, basicamente considérase que hai contaminación por ADN, e o ADN debe ser eliminado na fase inicial do experimento.
Por suposto, aínda hai algunhas situacións anormais, que se irán desglosando unha por unha a continuación.
3. Coñecementos avanzados
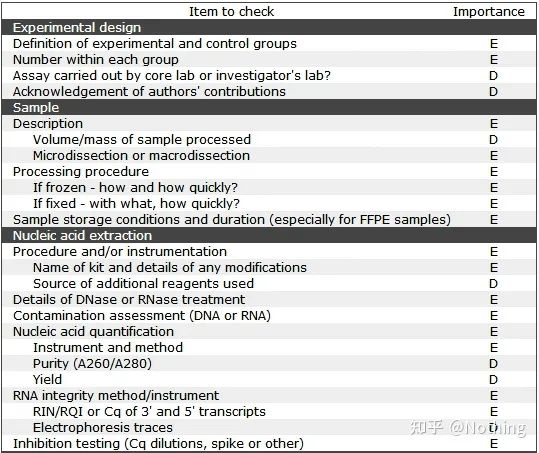
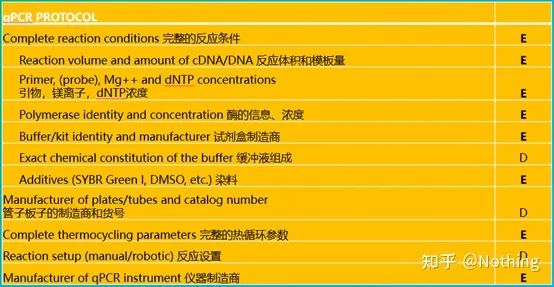
Para facer qPCR, teño que dicir MIQE,Información mínimapara Publicación deCuantitativoPCR en tempo realExperimentos: información mínima para publicar artigos sobre PCR cuantitativa en tempo realexperimentos.Para simplificar a comprensión de todos, simplificaremos o contido clave.
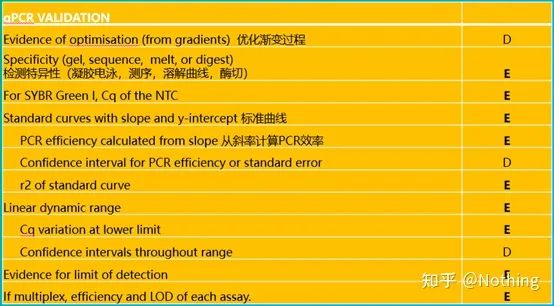
Podes buscar o texto orixinal de MIQE en Internet, e o máis importante é que estipula olista de verificación de datos que debe proporcionarse ao publicar un artigo .
Os revisores poden xulgar a calidade do experimento lendo estes detalles;os futuros lectores tamén poden usar isto para repetir ou mellorar o experimento.
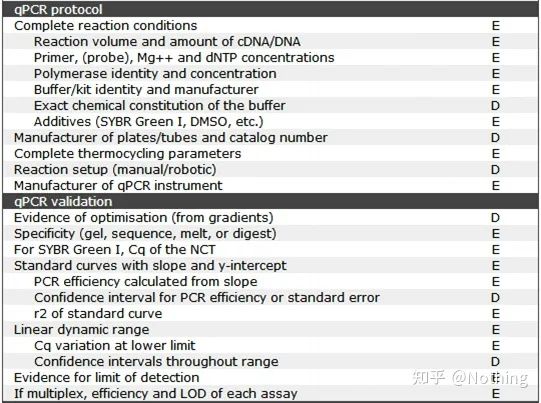
Cabe destacar que nesta lista, a importancia de cada lista está marcada con E ou D respectivamente.Qué significa?E: información esencial (debe presentarse);D: información desexable (proporcionar o máximo posible).
MIQE (1)—Deseño Experimental
Moitos canallas que remataron a súa defensa despois de rematar os seus estudos de posgrao non saberán deseñar un experimento de forma independente, abrir os seus cadernos e facer o que lles di o profesor.Como resultado, o deseño experimental non foi rigoroso e o departamento editorial da revista dixo que querían maquillar esta imaxe e esa imaxe, polo que o fixeron atónitos.¡Así se fan os cabróns!
Máis preto da casa, o primeiro principio do experimento é determinaro rigor da lóxica experimental.O máis fundamental é o deseño experimental, e o máis importante sobre o deseño experimental é como establecer a mostra obxectivo, a mostra de referencia (control) e o número de repeticións, para que os datos experimentais poidan ser referenciados, comparables e convincentes.
A mostra obxectivorefírese á mostra que nos obriga a detectar o xene diana despois dun determinado tratamento.A mostra de referenciaé a mostra sen ningún tratamento, que en bioloxía adoita denominarse tipo salvaxe.
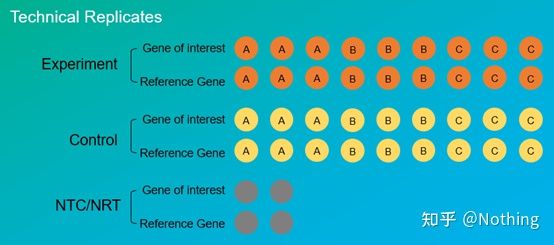
Réplicas experimentaisson moi importantes.Xeralmente, o número de réplicas persuasivas debe ser superior a tres.Hai que distinguir o que é a replicación biolóxica e o que é a replicación técnica.
Réplicas biolóxicas: O mesmo experimento de verificación feito con diferentes materiais (tempo, plantas, lotes, placas de reacción).
Duplicación biolóxica
Poñamos como exemplo o tratamento con pesticidas da pementa.Queremos pulverizar pesticidas nas tres plantas de ABC, entón as tres plantas de ABC son tres réplicas biolóxicas, e son o mesmo experimento de verificación realizado con materiais diferentes.Pero como experimento, definitivamente é necesario un control, polo que podemos pulverizar unha das ramas da planta A para formar un grupo experimental da planta A, e non pulverizar as outras ramas da planta A para formar un grupo de control.Fai o mesmo para B e C.
Réplicas técnicas (réplicas técnicas): É un experimento repetido deseñado para evitar erros causados polo funcionamento, que en realidade é un burato duplicado incluído no mesmo material.Tanto os tratamentos como os controis deben ter configuracións replicadas (mínimo tres) do xene diana e do xene de referencia interno.
Repetición técnica
Tome de novo como exemplo o pemento tratado con pesticidas.Para o grupo experimental da planta A, fixemos tres buratos de PCR de 1, 2 e 3 para o seu xene diana e o xene de referencia interno respectivamente, para tomar a media despois da detección.Para o control da planta A tamén se tratan do mesmo xeito os Grupos A.Do mesmo xeito, fai o mesmo tratamento para as plantas B e C.Esta é a repetición técnica.
Paga a pena sinalar queo que entra nas estatísticas é a repetición biolóxica, e a repetición técnica é comprobar se hai fenómenos aleatorios no proceso experimental, para facer creíbles os resultados experimentais, é dicir, para evitar erros tomando a súa media como adoitamos dicir.
Controis negativos: NTC e NRT
NTC (Control sen plantilla), un control sen plantilla, úsase para verificar se o material experimental está contaminado.Xeralmente, a auga úsase como modelo.Se hai unha reacción fluorescente, indica que se produciu contaminación por ácidos nucleicos no laboratorio.
Estas contaminacións proveñen de: auga impura, reactivos non cualificados que conteñen ADN endóxeno, contaminación de cebadores, contaminación de equipos de laboratorio, contaminación de aerosols, etc., necesidade de usar eliminadores de RNase e inhibidores de RNase.A contaminación por aerosol é a máis difícil de atopar.Imaxina que o teu laboratorio é como o smog, con varios ácidos nucleicos suspendidos no aire.
NRT (sen transcriptase inversa), o control sen transcrición inversa, é o ARN transcrito non inversamente como control negativo, que é o control do residuo de ADNg.
Ao facer a expresión xénica, a cantidade de ARN detéctase detectando a cantidade de ADNc despois da transcrición inversa.Se hai residuo de ADNg cando se purifica o ARN, provocará erros nos resultados experimentais, porque os resultados reais obtidos son ADNg e ADNc.A nivel agregado, non só o ADNc, o ADNg debe ser eliminado completamente durante a extracción de ARN.
MIQE (2): información de mostra
A chamada información de mostra significa que cando publicamos un artigo sobre qPCR, debemos explicar a información da mostra con claridade, que é unha parte indispensable do artigo.Do mesmo xeito, cando procesamos mostras, tamén debemos regular as nosas propias operacións para garantir a validez das mostras.
A descrición da mostra é só un resultado, e debemos prestar máis atención aos materiais tomados durante todo o experimento.
Selección de materiais experimentais
Mostras de sangue: escolla sangue fresco, non máis de 4 horas.Mostras celulares: escolle recoller células frescas nun período de crecemento vigoroso.Tecido animal: escolla tecido fresco e en crecemento vigoroso.Tecido vexetal: escolle tecido novo e fresco.
Debes ter notado que hai unha palabra clave nestas poucas frases: fresco .
Para as mostras anteriores, o kit mellor, rendible e estable do mercado é o kit de Foregene, que pode extraer rápida e facilmente o seu ADN e ARN.
Kit de illamento de ARN total celular
Kit de illamento de ARN total animal
Kit de illamento de ARN total da planta
Kit de illamento de ARN total de plantas Plus
Kit de illamento de ADN vexetal
Almacenamento de materiais experimentais
En xeral, non recomendamos almacenar mostras, se as condicións o permiten.Non obstante, hai moitos amigos que non poden realizar experimentos inmediatamente despois da toma de mostras, e algúns incluso precisan levar tanques de nitróxeno líquido ao campo para a toma de mostras.
Para este tipo de amigo traballador, só podo dicir que non entendes os consumibles de reactivos.Agora moitas empresas de consumibles de reactivos producen reactivos que poden almacenar mostras de ARN a temperatura ambiente, e pode optar por usalos.O método de almacenamento convencional é o almacenamento de nitróxeno líquido, utilizando un pequeno tanque de nitróxeno líquido que é fácil de transportar.Despois de levar a mostra ao laboratorio, gárdaa nun frigorífico a -80 °C.
Para experimentos que impliquen ARN, débese seguir o principio de seis palabras:baixa temperatura, sen encimas,erápido .
O concepto de baixa temperatura é fácil de entender;sen encimas, a RNase está en todas partes do mundo no que vivimos (se non o VIH mataríase), así que como evitar a RNase ao facer experimentos é un concepto moi importante;rápido,Non hai Kung Fu no mundo que non se poida romper, só a velocidade non se pode romper.
Polo tanto, en certo sentido, canto menor sexa o tempo de extracción, mellor será o kit.Por que faiForexeneO kit fai fincapé na velocidade, porque o saben ben.
PD: Algunhas rapazas fan experimentos con moito coidado, pero non son tan boas como un slam dunk despois de varios anos de traballo.Eles senten que Deus é inxusto, queixándose dos demais e buscando a vida.De feito, ela non o entendía.Non protexeu ben o ARN e o xogador de slam dunk era áxil.Cando estaba facendo o experimento, pensou que remataría o slam dunk con tres veces, cinco veces e dúas divisións, pero fixo o experimento ben.
Nota: Máis lento, máis posibilidades de invasión da RNase.Como adestrarte para ser rápido?Non hai maneira, só practica máis.
Para diferentes experimentos e diferentes mostras, aínda é necesario ler máis literatura e escoller un método axeitado para o procesamento.Para o proceso de recollida e almacenamento de mostras, MIQE require que estea claramente escrito no documento, para que os revisores poidan revisar a fiabilidade do documento, e tamén é conveniente que os mozos abraiados repitan o seu experimento.
Aínda que os experimentos biolóxicos son difíciles, son de alta gama.Se non tes coidado, podes derrubar o mundo.Por exemplo, converter o SARS nunha crise bioquímica ou facer arroz híbrido para salvar a 1.300 millóns de persoas.A imaxe de abaixo é un experimento químico, debes entender o orgulloso que estás da túa investigación só mirando a súa aparencia de pau.Esquéceo, non o fagas negro.
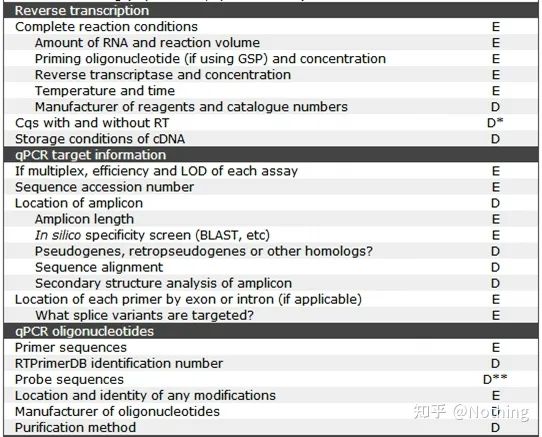
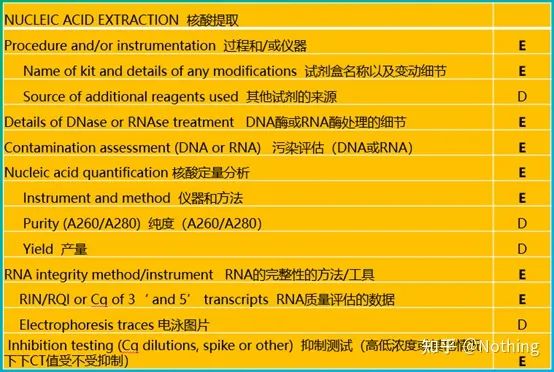
MIQE (3) – extracción de ácidos nucleicos.
A extracción de ácidos nucleicos é un gran evento, e todos os experimentos de bioloxía molecular comezan coa extracción de ácidos nucleicos.En primeiro lugar, copiemos o contido do MIQE sobre a extracción de ácidos nucleicos.
Mirando este formulario, non podes quedarte na superficie.A forma é un dogma.Para ser un estudante superior, debes preguntar por que.O contido esencial desta táboa é: Perseguira pureza, integridade, consistencia e cantidade de extracción de ARN .
A primeira parte doproceso ou instrumento é a etapa de extracción do ácido nucleico.Se usa un extractor automático de ácidos nucleicos para extraer (avanzado, póñase en contacto comigo para a compra), cómpre indicar o nome do modelo do instrumento.
O nome do kit e
Que kit se utilizou para os detalles do cambio, que reactivos especiais se engadiron ou que operacións especiais se fixeron deben explicarse claramente para que outros poidan repetir facilmente o seu experimento.
Algunhas persoas engaden algúns reactivos especiais á hora de extraer mostras especiais, pensando que esta é a súa arma secreta e non se lles conta aos demais.Aínda que o manteñen en segredo, tamén perden a oportunidade de facer brillar o teu artigo.Non sexas intelixente, tes que ser máis honesto que o vello Zhang na investigación científica, se queres ser intelixente, o artigo converterache en estúpido.
debe lembrar o número de produto do kitcando encargas o kit e escribes o artigo.Xeralmente hai dous números no kit: Cat: número de catálogo (número de produto, número de artigo), Lote: número de lote de produto (Usado para indicar de que lote procede o produto).
Ademais, o número CAS utilízase a miúdo cando se solicitan reactivos bioquímicos, e vouno popularizar xuntos.O número CAS é o número que dá a American Chemical Society a cada novo medicamento químico.Xeralmente, tres números están conectados por un guión.Número CAS de Rushui: 7732-18-5.Os produtos químicos adoitan ter varios alias, pero o número CAS é único.Cando solicite un medicamento, primeiro pode comprobar o seu número CAS.
Máis preto de casa, por que temos que describir estas cousas con claridade?De feito, tamén é para comprobar a calidade da extracción de ARN.O uso de instrumentos e kits fará que a extracción de ARN sexa máis consistente.A escala de extracción dos laboratorios ordinarios non é grande e pódese obter con kits.
Os detalles do tratamento con DNase ou RNase
A cuestión importante da PCR cuantitativa fluorescente é evitar a contaminación do ADN e non experimentar se hai contaminación.Polo tanto, é imperativo indicar o proceso que utilizou para procesar o ADN, para demostrar que o ADN no proceso experimental foi eliminado por completo e completamente.representado por un diagrama esquemático.
Diagrama esquemático de ARN e ADN
En xeral, o método para eliminar o ADN é tratar o ARN con DNase despois da extracción.Non obstante, estes son métodos relativamente antigos.Os kits comerciais de extracción de ARN foron capaces de eliminar o ADN durante o proceso de extracción sen engadir DNase .Por exemplo, unha serie de kits de Foregene.
Nota: Eliminar ADN durante a extracción de ARN é unha arma de dobre fío moi perigosa, que prolongará o tempo de operación da extracción de ARN e aumentará o risco de degradación do ARN.Basicamente, é unha compensación entre o rendemento e a pureza de ARN.
Ademais, a cantidade de DNase engadida á columna de adsorción a base de sílice é moi pequena e hai que usar DNase de alta calidade para conseguir o efecto.A DNase non optimizada non se pode dixerir rápida e completamente.Esta é unha proba do nivel técnico do comerciante.Por suposto, hai aínda máis comerciantes estraños que presumen de que o ADN se pode eliminar sen DNase.Pódese dicir que quen presume de que o ADN pode ser eliminado completamente sen ADNase é un gamberro.O ADN é unha estrutura de dobre cadea relativamente estable e non se pode eliminar só falando e rindo.
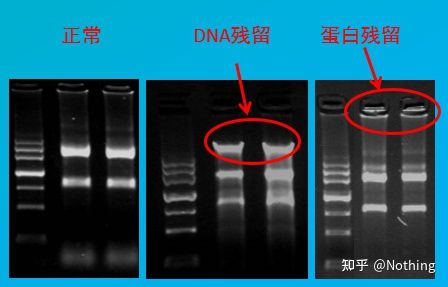
Avaliación da contaminación
método de avaliación: detección de electroforese, 1% de agarosa, 6V/cm, 15min, carga 1-3 ul
Análise cuantitativa de ácidos nucleicos
adoita medirse mediante un espectrofotómetro UV.Permítanme primeiro popularizar o significado dos tres valores de OD260, OD280 e OD230.
·OD260nm: é a lonxitude de onda de absorción do pico de absorción máis alto do ácido nucleico, e o mellor valor medido varía de 0,1 a 1,0.En caso contrario, dilúe ou concentre a mostra para situala dentro do rango.
·OD280nm: é a lonxitude de onda de absorción do pico de absorción máis alto de proteínas e substancias fenólicas.
·OD230nm: é a lonxitude de onda de absorción do pico de absorción máis alto de hidratos de carbono.
A continuación, imos falar sobre o papel de cada indicador.Para A260, pódese usar para medir o rendemento de ácido nucleico.Cando OD260=1, dsDNA=50μg/ml, ssDNA=37μg/ml, ARN=40μg/ml.
Para a pureza, necesitamos mirar as relacións que habitualmente vemos: OD260/280 e OD260/230.
·ADN puro: OD260/280 é aproximadamente igual a 1,8.Cando é superior a 1,9, indica que hai contaminación por ARN, e cando é inferior a 1,6, indica que hai contaminación por proteínas e fenol.
·ARN puro: 1,7
·OD260/230: se é ADN ou ARN, o valor de referencia é 2,5.Cando é inferior a 2,0, indica que hai contaminación de azucre, sal e materia orgánica.
Integridade do ARN
É moi importante medir a integridade do ARN.Xeralmente, é necesario facer un experimento de xel de desnaturalización de ARN para comprobar se o brillo entre o ARN 28S e 18S é unha relación dobre.Cando aparece a terceira banda 5S, significa que o ARN comezou a degradarse, excepto nos invertebrados.
Datos para a avaliación da calidade do ARN: Ademais das probas anteriores, tamén hai algunhas probas de instrumentos máis avanzadas en canto á integridade do ARN, como a proba de integridade RQI do sistema de electroforese automática Experion, que pode detectar se o ARN se degrada de forma invisible.
Na investigación científica, a PCR cuantitativa fluorescente é unha comparación entre o xene diana e o xene de referencia interno.Polo tanto, no proceso de preservación de mostras de ARN, extracción de ARN, etc., o obxectivo principal é garantir a integridade do ARN.
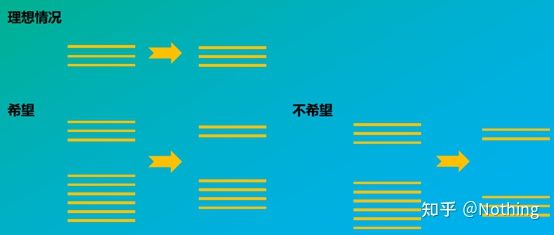
Como a integridade do ARN afecta o equilibrio entre o xene diana e o xene de referencia interno pódese entender facilmente a partir da figura seguinte.A degradación levará á incompletitude do xene, xa sexa a incompletitude do xene de referencia interno ou a incompletitude do xene obxectivo, terá un gran impacto nos datos.
O diagrama esquemático do xene diana e do xene de referencia non debe ser certo
Proba de inhibición (se o valor CT se suprime en alta ou baixa concentración ou noutras condicións)
Tomando esta figura como exemplo, os valores de Ct das cinco curvas son os seguintes.A distribución dos valores de CT entre as curvas é desigual e os valores de Ct retrasan en concentracións altas e baixas, como é o caso da inhibición da PCR.
Punto clave: no proceso de extracción de ARN, cómpre abandonar as ideas erróneas e establecer outras correctas.
A idea equivocada é: a extracción de ARN só persegue o rendemento, pensando que canto maior sexa a cantidade de ARN obtida, mellor.De feito, cando facemos cuantificación, se o número de xenes non é moi grande, non necesitamos moito ARN.A cantidade de ARN que extraes é máis que suficiente.
O concepto correcto é:A extracción de ARN debe buscar pureza, integridade e consistencia.A pureza pode garantir que a posterior transcrición inversa non se inhiba e que os datos non se verán afectados polo ADN.A integridade garante o equilibrio das secuencias obxectivo e das referencias internas.A consistencia garante unha carga estable da mostra.
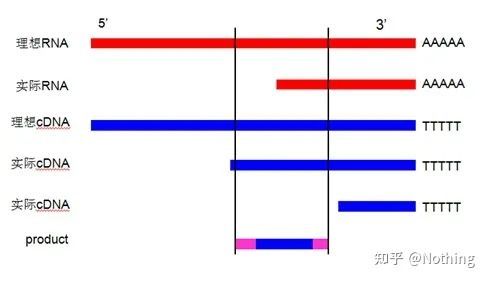
MIQE (4) – transcrición inversa
Equívoco: a procura dun maior volume de mostra.
Concepto correcto: Busca a consistencia (estabilidade), independentemente da cantidade de ARN cargado, a eficiencia da transcrición inversa segue sendo consistente, garantindo que as diferenzas no ADNc poidan reflectir verdadeiramente as diferenzas no ARNm.
Explicamos este proceso cun diagrama esquemático:
Diagrama esquemático da eficiencia da transcrición inversa, non é certo
En primeiro lugar, necesitamos comprender a diferenza entre o proceso de transcrición inversa e o proceso de PCR.A PCR sofre múltiples procesos de quecemento e recocido, e o fragmento obxectivo crece exponencialmente;mentres que a transcrición inversa non ten este proceso, podemos imaxinar que a transcrición inversa é realmente un a un Durante o proceso de replicación, tantas pezas de ARN
como hai pode obter tantas pezas de información de ADNc, debería entenderse a estas alturas, porque os fragmentos grandes e pequenos foron transcritos inversamente e é imposible centrarse nun fragmento.E debido a que a cantidade de ARN é relativamente pequena, a cantidade de ADNc obtido tamén é relativamente pequena, a diferenza da PCR, que ten un efecto de amplificación, polo que é basicamente imposible de detectar.
Resultados da electroforese de ADNc
En segundo lugar, idealmente, a transcrición inversa realízase un a un, pero ningunha transcriptase inversa de ningunha empresa pode conseguir este efecto.Basicamente, a eficiencia da maioría das transcriptases inversas vaga entre o 30-50%.Se este é o caso, preferiríamos ter unha eficiencia de transcrición inversa relativamente estable, que é o que queremos ver na figura: 3 ARN obteñen 2 cDNA, 6 ARN obteñen 4 cDNA, polo que non importa canta mostra se cargue, a eficiencia da transcrición inversa é relativamente estable.Non queremos ver a situación na que a eficiencia da transcrición inversa é inestable e se inhibe a alta concentración.
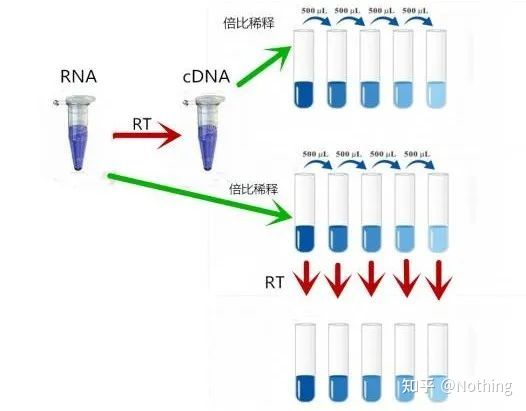
Entón, como verificar se a eficiencia da transcrición inversa é estable?O método é moi sinxelo, só tes que facer unha proba de comparación: unha é a transcrición inversa en cDNA despois de duplicar a dilución de ARN, e a outra é facer a duplicación de dilución tras a transcrición inversa en cDNA, e despois facer qPCR para ver a pendente obtida ¿É consistente?Como estudante superior, debes entendelo en segundos.Como se mostra a continuación:
Dilución de ARN e ADNc para comprobar se a eficacia da transcrición inversa é estable
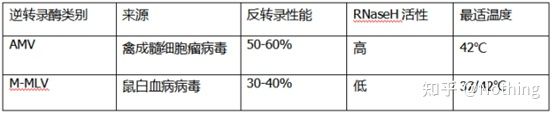
Transcriptase inversa e kit
Como pode a PCR cuantitativa fluorescente perfecta ter unha excelente transcriptase inversa e un kit?A transcriptase inversa divídese aproximadamente en dous tipos segundo a fonte, AMV ouM-MLV, e o seu rendemento é o mesmo que o mostrado na táboa.
Actividade da RNase H
A RNase H é Ribonuclease H, o nome chinés é ribonuclease H, que é unha endoribonuclease que pode hidrolizar especificamente o ARN na cadea híbrida ADN-ARN.A RNase H non pode hidrolizar os enlaces fosfodiéster en ADN ou ARN monocatenario ou bicatenario, é dicir, non pode dixerir ADN ou ARN monocatenario ou bicatenario.Úsase habitualmente na síntese da segunda cadea de ADNc.
É unha cousa estraña.Dicimos que a transcriptase inversa ten actividade RNase H, non que a transcriptase inversa conteña RNase H, e é posible que non sexa posible separar a RNase H da transcriptase inversa, quizais pola conformación de certos grupos na transcriptase inversa. Esta actividade é causada pola transcriptase inversa.
Polo tanto, independentemente da maior eficiencia de transcrición inversa do AMV, a súa actividade RNase H reduce o rendemento de cDNA.Por suposto, os fabricantes de reactivos optimizan constantemente os seus produtos para eliminar o máximo posible a actividade da RNase H na transcriptase inversa para aumentar o rendemento de ADNc.
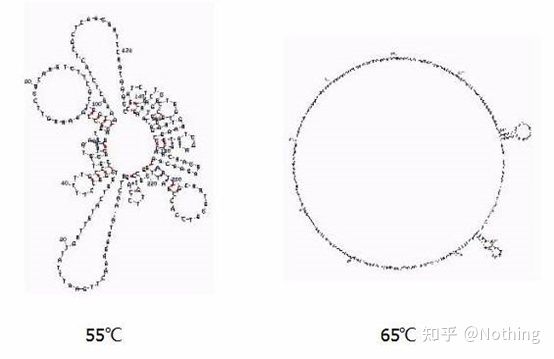
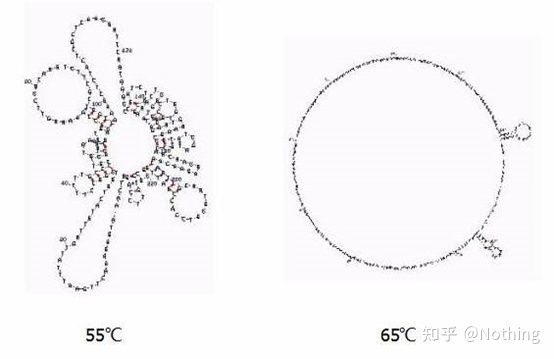
Temperatura de recocido
Estrutura secundaria do ARN a diferentes temperaturas
Vexa a figura anterior para ver a estrutura secundaria do ARN a diferentes temperaturas e use a ferramenta en liña mFold para determinar a estrutura secundaria do fragmento obxectivo en condicións específicas de temperatura e concentración de sal.A 55 °C, a estrutura secundaria do ARN aínda é moi complexa, a transcriptase inversa non pode funcionar e a estrutura secundaria non pode resolverse completamente ata 65 °C, mentres que a temperatura óptima de AMV e M-MLV é moito máis baixa que esta temperatura.
que facer?A estrutura secundaria é o emparellamento complementario do propio molde, o que leva a unha forte competencia entre o cebador e a transcriptase inversa e o molde, dando lugar a unha serie de problemas como a baixa E e a mala repetibilidade.
que facer?Só aumenta a temperatura de recocido o máximo posible.
Moitos fabricantes de reactivos están a mellorar a súa transcriptase inversa mediante a enxeñaría xenética.Algúns aumentan a temperatura de reacción, como Jifan e Aidelai, e outros eliminan o grupo activo do encima RNase H para mellorar a afinidade entre o encima e o molde de ARN.A alta afinidade pode espremer competitivamente a estrutura secundaria e ler sen problemas, e tamén mellorar moito a eficiencia da transcrición inversa.
Punto clave: a transcrición inversa é máis importante para buscar a coherencia da eficiencia da transcrición inversa (as encimas non só deben ser eficientes senón tamén estables), en lugar da cantidade de mostra cargada, se non se trata dunha PCR cuantitativa fluorescente especialmente a gran escala, non será posible en absoluto.ADNc múltiples.
Varios fabricantes tamén fixeron algúns esforzos na procura da coherencia.Por exemplo, a maioría das empresas agora empaquetaron a transcrición inversa como un kit estándar para a venda, o que é unha boa opción.
Por exemplo, os kits da serie RT Easy de Foregene:
RT Easy I (Premestura mestra para o kit de síntese de ADNc da primeira cadea)
MIQE (5) - información do xene obxectivo
A figura anterior explica
1. Se este xene é efectivo para experimentos repetidos, xeralmente pódese verificar mediante experimentos repetidos.
2. ID xene, xa sabes.
3. A lonxitude do xene, a lonxitude total do xene obxectivo definitivamente non é ningún problema.Ao deseñar cebadores, asegúrese de que a lonxitude do amplicón estea entre 80 e 200 pb para garantir unha mellor eficiencia de amplificación.
4. Información de comparación de explosión de secuencias, o xene obxectivo debe ser comparado no banco de xenes para evitar a amplificación inespecífica.
5. Presenza de pseudoxenes.Un pseudoxene é unha secuencia de ADN similar a un xene normal pero perde a súa función normal.A miúdo existe na familia multixenética dos eucariotas.Adoita representarse por ψ.É unha copia de ADN xenómico non funcional no xenoma que é moi semellante á secuencia do xene codificador., xeralmente non se transcriben e non teñen un significado fisiolóxico claro.
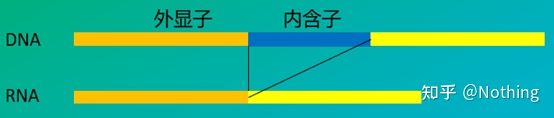
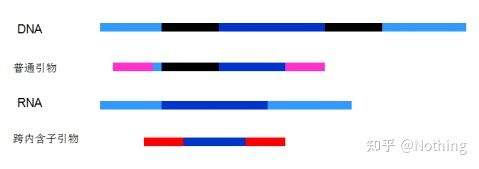
6. Posición dos cebadores en relación aos exóns e intróns.Nos primeiros anos, cando resolvemos o problema da contaminación do ADN, adoitamos prestar atención ás posicións dos cebadores, exóns e intróns, e en xeral consideramos deseñar cebadores entre intróns para evitar a amplificación do ADN.Vexa a seguinte figura: o negro representa intróns, varios azuis representan exóns, o rosa representa cebadores comúns e o vermello brillante representa cebadores que abarcan intróns.
Esquemático, nunca certo
Que plan perfecto parece, pero de feito, na maioría dos casos, os cebadores trans-intróns non son tan máxicos como se imaxinaba, e tamén provocarán unha amplificación inespecífica.Polo tanto, a mellor forma de evitar a contaminación do ADN é eliminar completamente o ADN.
7. Predición da conformación.Usando este exemplo de novo, use a ferramenta en liña mFold para determinar a estrutura secundaria do fragmento obxectivo a unha temperatura e concentración de sal específicas.
Estrutura secundaria do ARN a diferentes temperaturas
A estrutura secundaria é o emparellamento complementario do propio modelo, o que levará a unha forte competencia entre o emparellamento do cebador e do modelo, e as posibilidades de unión do cebador son menores, o que orixina unha serie de problemas como a baixa E e a mala repetibilidade.A través da predición de software, se non hai un problema de estrutura secundaria, sería xenial.Se o hai, o noso artigo de seguimento discutirá especificamente como resolver este problema.
MIQE (6) - Oligonucleótidos qPCR
Para a PCR cuantitativa fluorescente, o primeiro co que loitas todos os días é a extracción de ARN, e o segundo pode ser o deseño de cebadores.
En primeiro lugar, aínda comprobamos as regras sobre o deseño de imprimacións segundo a lista de verificación do MIQE.É tan sinxelo que os cabróns poden rir, e podemos rematalo nunha frase: descubre a secuencia e a posición da sonda cebadora e o método de modificación.Para o método de purificación de cebadores, a síntese de cebadores é tan barata na actualidade, qPCR é digno de PAGE e métodos de purificación superiores, e a información do instrumento de síntese non é importante.Moita xente leva décadas facendo cebadores e non sabe que o sintetizador é ABI3900.
Respecto dos principios do deseño de cartillas, non tes que memorizalos de memoria, porque a maioría dos programas de deseño de cartillas ou ferramentas en liña poden facerse cargo destes problemas (ferramenta en liña recomendada primer3.ut.ee/), e o 99,999% do deseño de cartillas non se fai manualmente Mira, o autor ás veces deseña centos de cartillas ao día, se le un por un, quedará cruzado.
Só tes que comprobar os seguintes puntos despois de deseñados os imprimadores:
1. Deseñar cebadores preto do extremo 3′: no caso de usar cebadores oligo dT para a síntese da primeira cadea de ADNc, tendo en conta a eficacia da transcrición inversa e a integridade do ARN, os cebadores deseñados deben deseñarse preto do extremo 3′ para mellorar a eficiencia da amplificación.Use unha imaxe para explicar o seguinte (non hai forma de entender isto):
Por que os cebadores deberían deseñarse preto do extremo 3′, non debe ser certo
2. Valor TM: o valor Tm está a 55-65 °C (porque a actividade exonuclease é a máis alta a 60 °C), e o contido de GC está no 40%-60%.
3. BLAST: para evitar a amplificación inespecífica do xenoma, débese utilizar Blast para a verificación complementaria.
MIQE(7)—proceso de qPCR
1. Kit de qPCR
Segundo os requisitos de MIQE, debemos describir claramente as condicións de reacción completas no artigo, incluíndo a configuración do sistema de reacción de PCR, que kit se usa, quen é o fabricante, o tamaño do sistema de reacción, se se usa o método de tintura ou o método de sonda, a configuración do programa de PCR.Os condutores veteranos definitivamente descubrirán que, sempre que se seleccione o kit, a información anterior determínase basicamente.
Na actualidade, a fabricación e produción de kits de PCR cuantitativos fluorescentes é unha tecnoloxía moi madura.Mentres non elixas fabricantes moi malos, a probabilidade de problemas non é alta, pero aínda queremos compartir contigo algúns puntos:
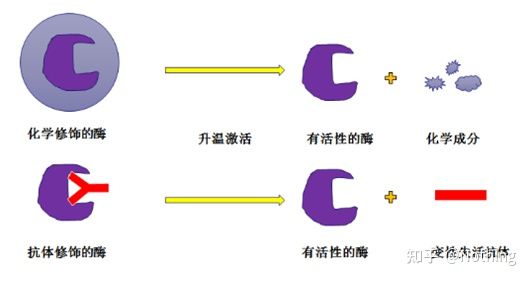
Enzima Taq de inicio en quente:A parte máis importante da PCR é o encima Taq de inicio en quente.As enzimas de inicio en quente no mercado xeralmente divídense en dous tipos, unha delas é unha enzima de inicio en quente modificada químicamente (pode imaxinala como incorporación de parafina) e a outra é unha enzima de inicio en quente para a modificación de anticorpos (unión antíxeno-anticorpo).A modificación química é unha das primeiras formas de iniciar encimas en quente.Cando se alcanza unha determinada temperatura, a enzima liberará a súa actividade.O encima de inicio en quente modificado con anticorpos utiliza métodos biolóxicos para bloquear a actividade do encima.Cando se alcanza unha determinada temperatura, o anticorpo será desnaturalizado e inactivado como proteína, e porase en xogo a actividade enzimática.
Non obstante, para que serve isto?Este é o caso, a actividade de liberación das encimas modificadas con anticorpos é máis rápida que a das encimas modificadas químicamente, polo que en termos de sensibilidade, as encimas modificadas con anticorpos teñen unha lixeira vantaxe, polo que basicamente non hai encimas modificadas químicamente nos kits do mercado.Se o hai, entón a tecnoloxía deste fabricante aínda está atrapada na era do milenio.
Concentración de ión magnesio:A concentración de ións magnesio é moi importante na reacción de PCR.A concentración adecuada de ións magnesio pode promover a liberación da actividade enzimática Taq.Se a concentración é demasiado baixa, a actividade enzimática reducirase significativamente;se a concentración é demasiado alta, mellorarase a amplificación non específica catalizada por encimas.A concentración de ións magnesio tamén afectará ao recocido dos cebadores, á temperatura de fusión do molde e aos produtos da PCR, afectando así o rendemento de fragmentos amplificados.A concentración de ións magnesio é xeralmente controlada a 25 mM.Por suposto, para un bo kit, a concentración de ións magnesio debe estar ben controlada.Algúns comerciantes engaden un axente quelante de ións de magnesio ao reactivo, o que pode lograr o efecto de axuste automático da concentración de ións de magnesio.
Concentración de colorante fluorescente:O colorante fluorescente, que é o SYBR Green que usamos habitualmente, xera fluorescencia principalmente ao unirse ao suco menor do ADN de dobre cadea, porque a unión do colorante ao ADN de dobre cadea é inespecífica, é dicir, mentres se combine con el ADN de dobre cadea, pode producirse fluorescencia, polo que os dímeros-cebadores combinarán os sinais de fondo co sistema para formar un patrón de ADN.
PS: Debido ás súas propiedades sensibles á luz, os produtos no mercado xeralmente envasan en tubos de centrífuga marróns opacos (como se mostra na imaxe de abaixo).Non obstante, isto atopará un problema.É difícil ver se o líquido é aspirado ao tomar a mostra.Neste sentido, Qingke é realmente o máis fácil de usar (como se mostra na imaxe de abaixo) e o tubo transparente está empaquetado nunha bolsa de lata opaca.Despois méteo nunha bolsa de lata, tendo en conta a comodidade de evitar a luz e a toma de mostras.Debes escoller o número de produto correcto.TSE204 é unha existencia súper rendible, o que me fai querer plantar herba.
Tamén é moi importante a concentración do colorante fluorescente.Se a concentración é demasiado baixa, a curva de amplificación non subirá na fase posterior e non é perfecta;se a concentración é demasiado alta, provocará interferencias de ruído.Dado que a PCR cuantitativa fluorescente depende principalmente do valor CT, se a concentración do colorante fluorescente non se axusta correctamente, o punto baixo é mellor que o punto alto.Por suposto, a concentración de colorante adecuada é a mellor.
ROX: Os colorantes ROX úsanse para corrixir os erros de sinal de fluorescencia de pozo a pozo.Algúns fabricantes de instrumentos requiren calibración, mentres que outros non.Por exemplo, o uso do instrumento de amplificación de PCR en tempo real de Thermo Fisher Scientific adoita requirir unha calibración, incluíndo 7300, 7500, 7500Fast, StepOnePlus, etc. As instrucións xerais do kit describiráno.
O qPCR Mix de Foregene tamén contén colorante ROX, que é conveniente para o seu uso en varios modelos.
Kit de PCR en tempo real-Taqman
Tratamento de enlaces de hidróxeno débil: O tratamento de enlaces de hidróxeno débiles é unha cuestión relativamente técnica.Nada leu os manuais de moitos kits, pero ningún deles mencionou este tema.De feito, é tan importante.A combinación de bases depende principalmente da forza dos enlaces de hidróxeno.Os enlaces de hidróxeno fortes son unha amplificación normal, e os enlaces de hidróxeno débiles conducen a unha amplificación inespecífica.Se os enlaces de hidróxeno débiles non se poden eliminar ben, non se pode evitar a amplificación inespecífica.No ámbito do autor, só unhas poucas empresas notaron este problema.Cando compre o kit, pode consultar se considerou unha solución a este respecto para o kit que quere escoller.
Volume de reacción: O sistema 20-50ul úsase máis habitualmente e é probable que volumes máis pequenos causen erros.En xeral, as instrucións do kit recomendarán o uso de volumes de reacción de PCR.Non sexa intelixente e use volumes máis pequenos para aforrar custos.o obxectivo de.O volume recomendado polos comerciantes realmente foi probado, e pode ser que non poidan resolver o problema dos erros causados por pequenos volumes.
2. O fabricante e o número de artigo da placa do tubo
Todo o mundo coñece o principio da PCR cuantitativa fluorescente.A recollida de fluorescencia realízase principalmente a través de tapas de tubos de PCR.Ao escoller consumibles de PCR, preste atención a dous puntos: boa transmisión de luz e axeitado para o instrumento.En xeral, as placas e tubos das principais marcas están ben, pero hai que escoller coidadosamente en canto a adaptación, se non, non poderás usar o instrumento.
4. Coñecementos de nivel superior
MIQE (8): validación de qPCR
Esta é a principal prioridade de qPCR!Tantos heroes caeron na area aquí.Por suposto, tamén é posible que teñas sorte e que os xenes que estudaches sexan sinxelos, polo que flotaches pola cova de xeo ao longo do vento.A información de verificación de qPCR ten como finalidade probar a fiabilidade dos datos.Enumeramos a información de verificación necesaria do seguinte xeito:
1.Proba de especificidade
A especificidade da amplificación do xene diana é probada comprobando se a imaxe de electroforese é unha soa banda;verificación de secuenciación;curva de fusión para ver se o mapa de picos é único;verificación da dixestión enzimática e outros métodos.
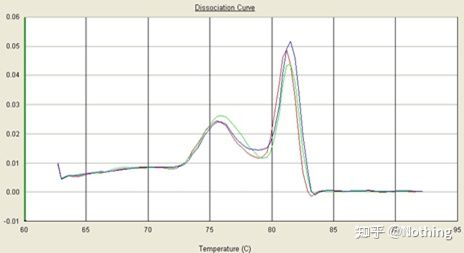
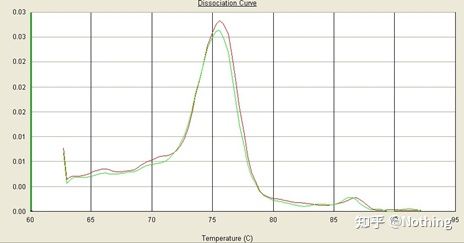
Aquí centrámonos en ta análise da amplificación inespecífica mediante o método das curvas de fusión.En xeral, cando deseñamos cebadores, o tamaño do fragmento do produto debe estar no intervalo de 80-200 pb, o que fai que a temperatura de fusión do produto PCR sexa de 80-85 °C.Polo tanto, se hai picos varios, debe haber outros produtos de amplificación non específicos;se o pico aparece por debaixo dos 80 °C, xeralmente considérase que é un dímero de cebador;se o pico aparece por riba dos 85 °C, xeralmente considérase contaminación por ADN ou máis Amplificación inespecífica de fragmentos grandes.
Nota: Ás veces só hai un único pico a 80 °C.Neste momento, este concepto debe ser respectado.É probable que os resultados da amplificación sexan todos dímeros de cebadores.
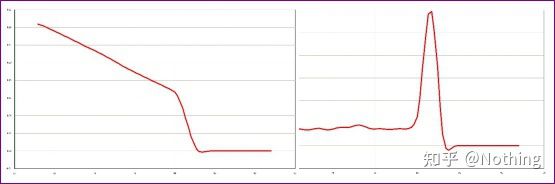
Curva de fusión normal (pico único sen amplificación inespecífica)
Curva de fusión problemática (amplificación inespecífica de picos espurios)
【Análise de casos】
Hai un pico principal, pero o dímero de imprimación é grave
A curva de fusión dun único pico da figura a continuación pode enganar facilmente os teus ollos, pensando que é un experimento perfecto, pero o resultado é completamente incorrecto.Neste momento, temos que mirar a temperatura de fusión.A temperatura máxima é inferior a 80 °C, o que é completamente dímero de imprimación.
Ningún fragmento obxectivo, todos os dímeros de cebador
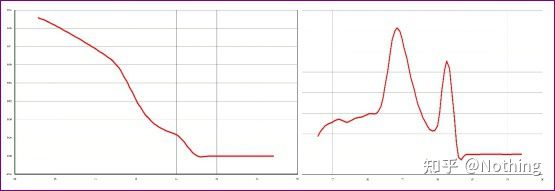
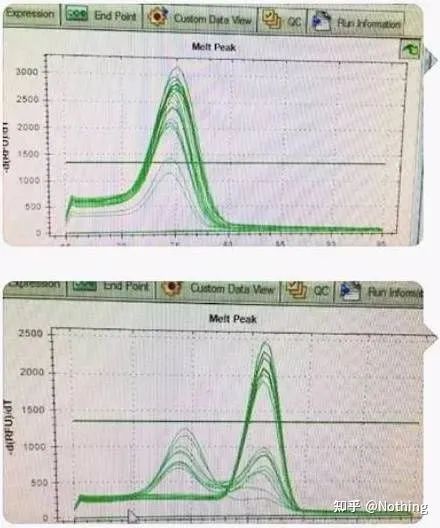
Aquí, o meu irmán non pode parar.A imaxe de abaixo é unha foto feita cun teléfono móbil que me enviou un canalla.Os reactivos que utilizou son marcas de uso habitual na industria.Cambiou dunha marca de prefixo T a outra marca de prefixo T.Creo que xa o adiviñaches.O bastón berroume: “O reactivo usado na primeira imaxe é demasiado bo e o pico é único.Máis tarde, despois de usar o reactivo que recomendaches, faise como a segunda imaxe, con picos mixtos.Fixéchesme desgraciado."
Separa as dúas gráficas.A primeira vista, un ten un único pico e o outro un dobre pico.Tonterías, un só pico está ben.É certo?
Peor que Dou E, se poño as dúas imaxes na imaxe de abaixo, entenderedes inmediatamente.De feito, estamos facilmente paralizados por este tipo de imaxes.Despois dunha análise coidadosa, descubrimos que: o pico da primeira figura está a 75 °C, que é completamente dímero de imprimación;o pico da segunda figura aparece a 75°C e 82°C, polo menos hai O produto aparece.
Imaxes de comentarios dos alumnos
Polo tanto, o problema fundamental non é o problema dos reactivos, senón o problema do deseño de imprimacións.Ao mesmo tempo, tamén proba que algunhas grandes marcas non son de calidade de ferro, e tamén proba o que dixo antes o meu irmán: Non é a marca de reactivos que apoia o teu artigo.É o teu artigo o que apoiou a marca de reactivos.Imaxinade, se o bastón non cambiase os reactivos, os datos incorrectos serían enviados á revista e o que pasaría sería unha traxedia.
2. Valor Ct do control en branco
Non explique, se o control en branco ten un valor Ct, non é contaminación?Non obstante, aínda debes comprender que control en branco ten un valor Ct.Se é NTC, significa que hai ADN estraño como contaminación de reactivos.Se é NRT, significa que o ARN extraído ten contaminación de ADN.
3. Curva estándar
Incluíndo a pendente e a fórmula de cálculo, a eficiencia da PCR pódese calcular mediante a fórmula.Un experimento perfecto require que a pendente da curva estándar se achegue a 3,32 e que R² se achegue a 0,9999.
4. Rango dinámico lineal
O rango dinámico da reacción é lineal.Segundo o modelo utilizado para xerar a curva estándar, o rango dinámico debe incluír polo menos 5 gradientes de concentración e prestar atención ao cambio dos valores de Ct en gradientes de concentración altos e gradientes de concentración baixos.
5. Precisión da detección
Os cambios nos resultados da qPCR, é dicir, a mala repetibilidade, é dicir, a mala precisión, son causados por moitos factores, entre eles a temperatura, a concentración e o funcionamento.A precisión da qPCR xeralmente faise menos controlable a medida que diminúe o número de copias.Idealmente, a variación dentro dos experimentos, esta variación técnica debería ser distinta da variación biolóxica, e as réplicas biolóxicas poden abordar directamente as diferenzas estatísticas nos resultados da qPCR entre grupos ou tratamentos.En particular, para os ensaios de diagnóstico, débese informar da mellor precisión entre ensaios (repetibilidade) entre sitios e operadores.
6. Eficiencia de detección e LOD (en qPCR múltiplex)
O LOD é a concentración máis baixa do 95% das mostras positivas detectadas.Noutras palabras, a concentración de LOD contida nun conxunto de réplicas de xenes diana non debe exceder o 5% das reaccións fallidas.Cando se realiza a análise de qPCR múltiple, especialmente para a detección simultánea de mutacións puntuais ou polimorfismos, a qPCR múltiple debe proporcionar probas de que a precisión de varios fragmentos diana non se ve comprometida no mesmo tubo, a detección múltiple e a detección en tubo único. A eficiencia e o LOD deben ser os mesmos.Especialmente cando se amplifican simultáneamente xenes diana de alta concentración e xenes diana de baixa concentración, hai que prestar atención a este problema.
Problemas e soluciónsEn xeral, os problemas que adoitan atoparse na depuración de qPCR céntranse nos seguintes aspectos:
·amplificación inespecífica
·Difícil elección da concentración de imprimación e problemas cos dímeros de imprimación
·A temperatura de recocido é imprecisa
·A estrutura secundaria afecta a eficiencia da amplificación
amplificación inespecífica
amplificación inespecíficaocorre, xeralmente considérase se o deseño de imprimación non é adecuado, pero se non tes présa por cambiar as imprimacións, podes probar primeiro os seguintes métodos (o principio tamén se adxunta):
·Aumenta a temperatura de recocido: intenta facer que os enlaces de hidróxeno débiles non poidan manterse;
·Acurtar o tempo de recocido e alongamento: reduce a posibilidade de enlaces de hidróxeno débiles;
·Reducir a concentración de cebadores: reduce a posibilidade de unión de cebadores redundantes e rexións non obxectivo;
Baixa eficiencia de amplificación
A situación oposta á amplificación non específica: baixa eficiencia de amplificación e as medidas para tratar a baixa eficiencia de amplificación son exactamente o contrario:
·Prolonga o tempo de recocido e alongamento;
·Cambia a PCR de tres pasos e reduce a temperatura de recocido;
·Aumentar a concentración de imprimación;
Ps: Moitos estudantes de posgrao nacidos nos anos 90 non están dispostos a estudar como depurar experimentos, e esperan que o kit poida resolver completamente o problema (se quere ir a unha empresa de reactivos para facer investigación e desenvolvemento despois da graduación), de feito, os fabricantes de reactivos tamén pensan deste xeito, espero que sexa un parvo. factores de absorción de enlaces.Para resolver facilmente o problema, os tolos aínda teñen que ler a introdución da compañía de reactivos para ver se hai algún factor que absorba os enlaces de hidróxeno débiles.
Difícil elección da concentración do cebador e problemas cos dímeros do cebador
Método 1: En xeral, as instrucións do kit para qPCR teñen sistemas recomendados e concentracións de cebador recomendadas.
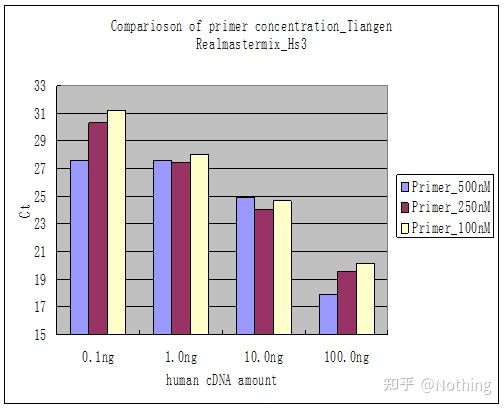
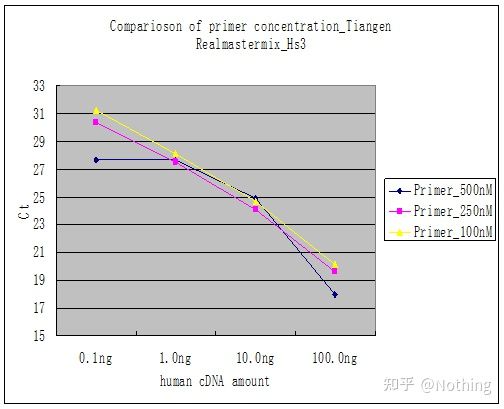
Método 2: Depuración configurando o gradiente de concentración do cebador.A imaxe de abaixo está roubada a unha empresa para ilustrala.A figura seguinte mostra os resultados cuantitativos de fluorescencia feitos con tres gradientes de concentración de cebadores (100 nM, 250 nM, 500 nM) e catro gradientes de concentración de moldes (0,1 ng, 1 ng, 10 ng, 100 ng).O valor Ct dos resultados experimentais está representado do seguinte xeito:
Selección da concentración de cebador Concatene cada concentración de cebador nunha liña do seguinte xeito:
A elección da concentración de cebador é obvia, a relación lineal da concentración de cebador de 100 nM e 250 nM é mellor e a relación lineal da concentración de cebador de 500 nM é relativamente pobre.En 100 nM e 250 nM, o valor Ct de 250 nM é relativamente pequeno, polo que a concentración óptima do cebador é de 250 nM.Na curva de fusión pódense observar xeralmente dímeros de imprimación severas.E se os cebadores deseñados non poden evitar os cebadores-dímeros?
Método 3: Reducir a cantidade de imprimacións e aumentar a temperatura de recocido (non hai que explicar).
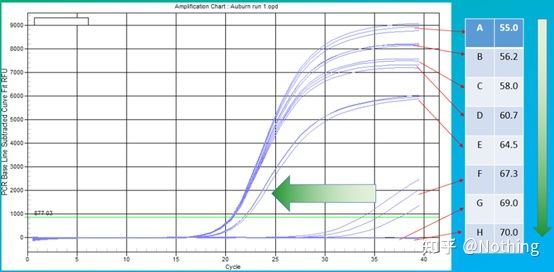
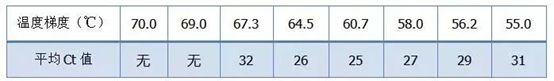
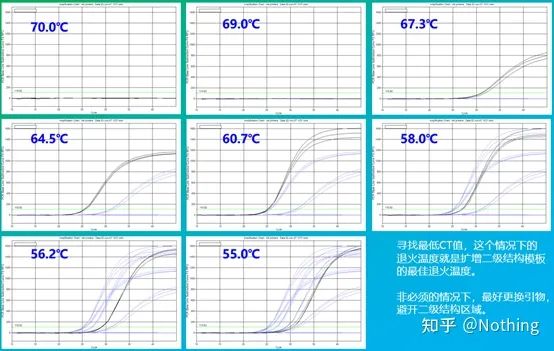
O valor empírico da temperatura de recocido é de 60 °C.Se non está seguro, como seleccionar unha temperatura de recocido máis adecuada?A resposta é a mesma que a elección da concentración de cebador:proba de gradiente.Fai unha foto da empresa Bio-rad para ilustrar o problema.Para a amplificación dun determinado fragmento obxectivo, establece oito gradientes de temperatura, cada un con tres repeticións, e a curva de amplificación obtida é a seguinte:
selección de temperatura de recocido:
·70°C, 69°C—Basicamente, os cebadores non se poden combinar, polo que non hai amplificación.
·67,3 °C: hai unha pequena cantidade de amplificación ao principio e o valor de Ct é relativamente grande.
·64,5°C——O valor Ct diminúe.
·A 60,7 °C, 58,0 °C, 56,2 °C e 55,0 °C, os valores de Ct tendían basicamente a ser estables, pero os valores finais de fluorescencia foron diferentes.
Como elixir?Principio: o primeiro principio é o valor Ct máis alto.Para o mesmo valor de Ct, escolla unha temperatura de recocido máis alta para evitar a dimerización e a amplificación inespecífica.Aínda que hai un valor de fluorescencia máis alto a 55 °C, pode haber nela dímeros ou amplificación inespecífica.
Pero se es tan intelixente coma ti, sen dúbida pensarás: loxicamente falando, se a reacción de PCR é moi específica, sempre que a concentración do cebador supere o requisito mínimo, os puntos alto e baixo non deberían ter ningún efecto, igual que os colorantes fluorescentes e os dNTP.De feito, sempre que a temperatura de recocido se optimice correctamente, o efecto da concentración de imprimación sobre o valor de Ct minimizarase naturalmente.
A temperatura de recocido optimízase correctamente e minimizarase o efecto da concentración de imprimación na TC
A estrutura secundaria afecta a eficiencia da amplificación
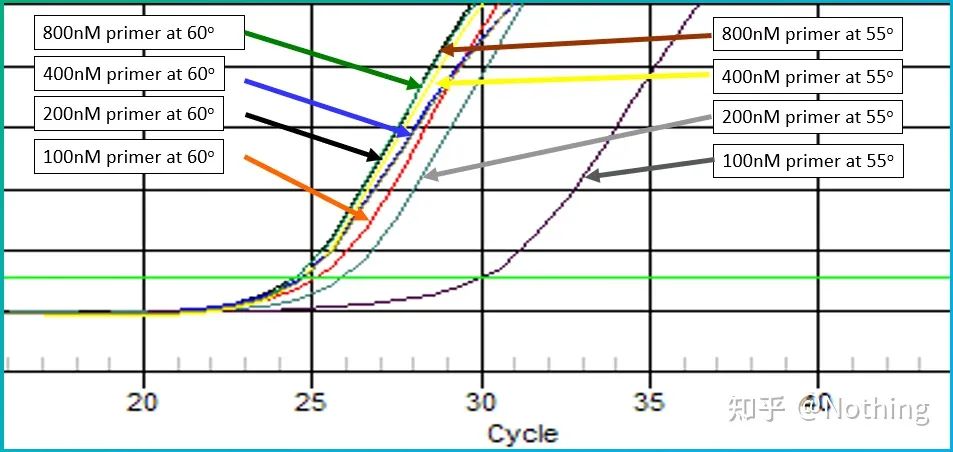
Saquemos a foto de Bio-rad para ilustrar o problema.Tamén deseña un gradiente de temperatura para amplificar un xene cunha estrutura secundaria.
Xorde a estrutura secundaria
Pódese ver que a medida que diminúe o gradiente de temperatura, comezan a aparecer produtos e o valor Ct avanza, acadando o valor mínimo a 60,7 °C, e despois a medida que diminúe o gradiente de temperatura, o valor Ct faise maior.Pola contra, a medida que aumenta a temperatura, ábrese a estrutura secundaria e aumenta a eficiencia de amplificación.Despois de alcanzar unha determinada temperatura, aumentar a temperatura non pode mellorar a eficiencia da amplificación.Porque os cebadores non se poden combinar de forma estable neste momento.Polo tanto,buscar a temperatura co valor Ct máis baixo, que é a mellor temperatura para amplificar o modelo de estrutura secundaria!Por suposto, os tolos intelixentes deben saber que, se non é necesario, o mellor é cambiar os cebadores e evitar a rexión da estrutura secundaria.
5. Nivel de aplicación
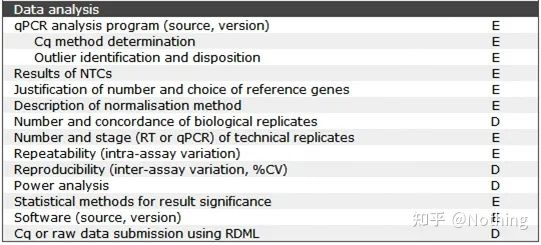
MIQE - Análise de datos
A análise de datos é dada principalmente polo instrumento de PCR cuantitativa fluorescente.No artigo anterior realizouse moito traballo de análise de datos, como o control en branco, que foi explicado no deseño do experimento.Aclaráronse os xenes de referencia internos, os números repetidos, etc., aquí explicamos principalmente a aplicación de qPCR.
A qPCR é moi utilizada, e a verificación experimental e o diagnóstico de ácidos nucleicos son os escenarios máis utilizados.
cuantificación absoluta
Log (concentración inicial) ten unha relación lineal co número de ciclos.Pódese trazar unha curva estándar a partir dun patrón cun número de copia inicial coñecido, é dicir, pódese obter a relación lineal da reacción de amplificación.Segundo o valor Ct da mostra, pódese calcular a concentración na mostra.A cantidade de modelos a incluír.
Método de cálculo cuantitativo absoluto
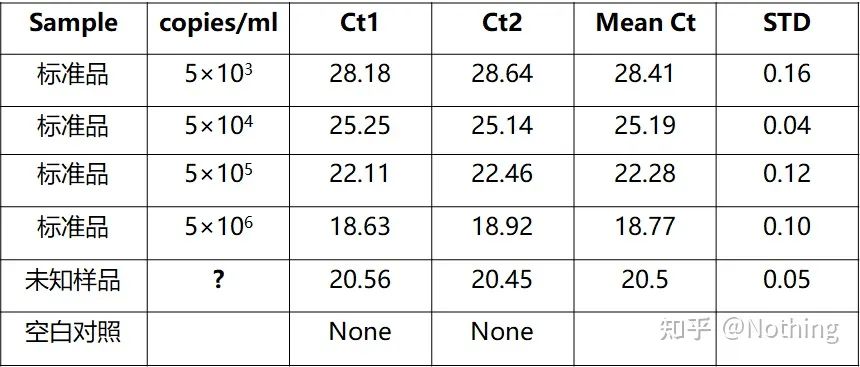
A cuantificación absoluta debe basearse na curva estándar.Para facer unha curva estándar, é necesario un estándar.Normalmente, o estándar é un plásmido obtido mediante a clonación do xene diana.Por que é un plásmido?Porque o ADN plasmídico circular é o máis estable.Diluír o produto estándar en 5 a 6 gradientes segundo a relación de duplicación (dilución de 10 veces) e prestar atención á uniformidade ao diluír.Deixa que o valor Ct caia entre 15-30.
Preparación estándar
Ao mesmo tempo, a mostra a probar tamén debe diluírse en consecuencia (recorde o factor de dilución), e o valor Ct tamén debe caer entre 15-30.O produto estándar + a mostra a probar colócanse na máquina xuntos.Despois da carreira, realizouse unha curva estándar coa substancia estándar, e as mostras a probar foron levadas á curva estándar para calcular a concentración.
A cuantificación do VHB do virus da hepatite B é unha cuantificación absoluta típica, que pode calcular o número de copias do virus en 1 ml de sangue.
Cálculo do número de copias
Concentración da mostra a probar (ng/ul) = DO260 × 50 ug/ml × factor de dilución
Peso molecular da mostra = número de bases × 324
O número de copia da mostra que se vai probar (copias/ul) = a concentración da mostra que se vai probar / o peso molecular da mostra × 6 × 1014
Método de cálculo do número de copias
O anterior é o método de cálculo para determinar a cantidade.Este é un problema matemático que se pode resolver despois de graduarse na escola secundaria, e os problemas matemáticos adoitan resolverse mediante ordenadores.Se non entendes, podes vir a comunicarte.
cuantificación relativa
A cuantificación relativa utilízase principalmente na investigación científica.Cantos virus hai en 1 ml de sangue, e é un virus de ADN, este é un evento relativamente determinista: pódese determinar a cantidade de sangue e o virus de ADN é relativamente estable.Non obstante, é difícil para nós comparar o número de copias de transcrición dun determinado xene nunha folla, porque é difícil determinar o tamaño, o peso e a tenrura da folla, a cantidade de ARN extraído é difícil de determinar e a eficiencia da transcrición inversa tamén é difícil de determinar, é dicir, calquera paso pode facer que os datos experimentais teñan erros e non se poidan usar.
Polo tanto, a cuantificación relativa debe introducir un elemento:o xen de referencia interno.
Noutras palabras, a cuantificación relativa é en realidade unha comparación entre o xene obxectivo e o xene de referencia interno.Comparado no mesmo tecido e na mesma célula, a influencia do tamaño da mostra, a cantidade de extracción de ARN, a eficiencia da transcrición inversa e a eficiencia da PCR é relativamente pequena.Debido ao pequeno tamaño da mostra, tanto os xenes de referencia internos como os xenes diana foron relativamente reducidos.É por iso que antes estivemos facendo fincapé na uniformidade e na estabilidade.
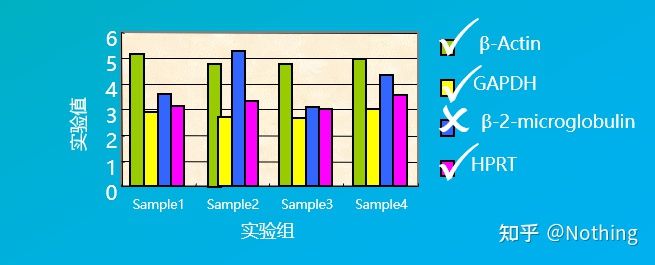
Os xenes de referencia internos son xeralmentexenes domésticos(xenes domésticos), que se refiren a unha clase de xenes que se expresan de forma estable en todas as células, e os seus produtos son necesarios para manter as actividades básicas da vida das células.
Non confunda este concepto.Os xenes domésticos son termos de función biolóxica, mentres que os xenes de referencia internos son termos técnicos experimentais.Os xenes domésticos deben pasar a validación antes de poder seleccionarse como xenes de referencia internos.
Por exemplo, seleccionamos varios xenes domésticos na figura seguinte para probar os seus niveis de expresión en diferentes células do tecido, e descubrimos que os niveis de expresión da β-2-microglobulina eran bastante diferentes dos dos outros tres xenes, polo que non se podían utilizar como xenes de referencia internos.
Despois de comprender a función de corrección do xene de referencia interno, derívanse dous algoritmos debido á introdución do xene de referencia interno.
·método de curva estándar dobre
·2 – Método △△Ct (método de comparación de valores CT)
Se estás interesado en estudar as funcións das especies e dos xenes, por favor, abandone a investigación sobre algoritmos e use fórmulas directamente, ou use máquinas directamente;se es un home heterosexual en matemáticas e enxeñería, non dubides.
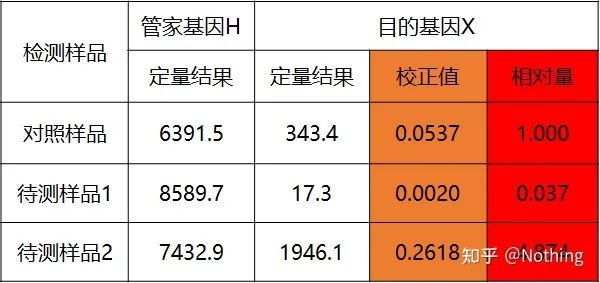
método de curva estándar dobre
Cuantifica o xene diana e o xene doméstico da mostra de control e da mostra que se vai probar a través da curva estándar, e despois calcula o valor relativo segundo a fórmula de cálculo, que é o nivel de expresión relativo.
Vantaxes: análise sinxela, optimización experimental relativamente sinxela
Desvantaxe: para cada xene, cada rolda de experimentos debe facer unha curva estándar
Aplicación: un dos dous métodos cuantitativos relativos máis utilizados e recoñecidos no estudo da regulación da expresión xénica.
A fórmula é a seguinte:
Os exemplos son os seguintes:
Calcula a cantidade relativa a partir do resultado cuantitativo
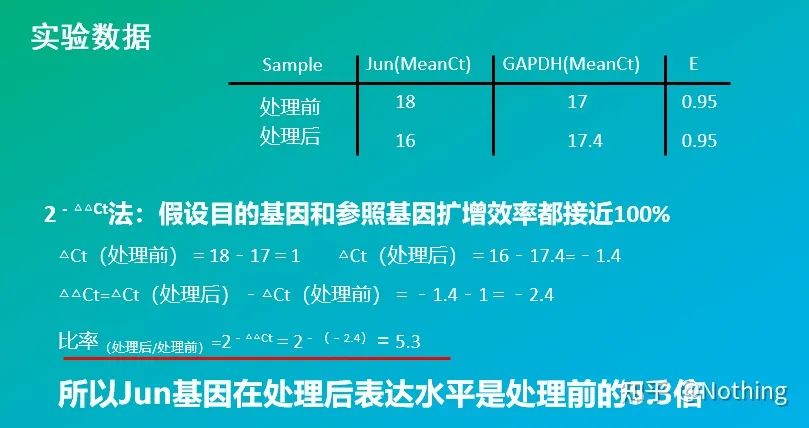
2 – Método △△Ct (método de comparación de valores CT)
Vantaxes: Non é necesario facer unha curva estándar
Desvantaxes: Suponse que a eficiencia de amplificación é próxima ao 100%;a desviación estándar é < 5%, e asúmese que a curva estándar e a eficiencia entre cada amplificación son consistentes;a optimización das condicións experimentais é máis complicada.
Aplicación: un dos dous métodos cuantitativos relativos máis utilizados e recoñecidos no estudo da regulación da expresión xénica.
Por suposto, a eficiencia da amplificación adoita ser imposible de ser perfectamente 1. Método de corrección: se sabemos que o xene obxectivo e o xene de referencia teñen a mesma eficiencia de amplificación, pero a eficiencia de amplificación non é igual a 1, entón 2-△△Ct pódese corrixir como: (1+E )-△△Ct, por exemplo, se pode calcular a eficiencia de amplificación 95 para corrixirse. 1,95 △△ Ct
Ata agora, o contido sobre a PCR cuantitativa fluorescente chegou ao seu fin.
Hora de publicación: abril-06-2023