As tres letras SNP son omnipresentes no estudo da xenética de poboacións.Independentemente da investigación de enfermidades humanas, o posicionamento dos trazos dos cultivos, a evolución animal e a ecoloxía molecular, os SNP son necesarios como base.Non obstante, se non tes un coñecemento profundo da xenética moderna baseada na secuenciación de alto rendemento e, fronte a estas tres letras, parece "o descoñecido máis familiar", entón non podes levar a cabo a investigación posterior.Entón, antes de emprender a investigación de seguimento, imos dar un ollo ao que é SNP.
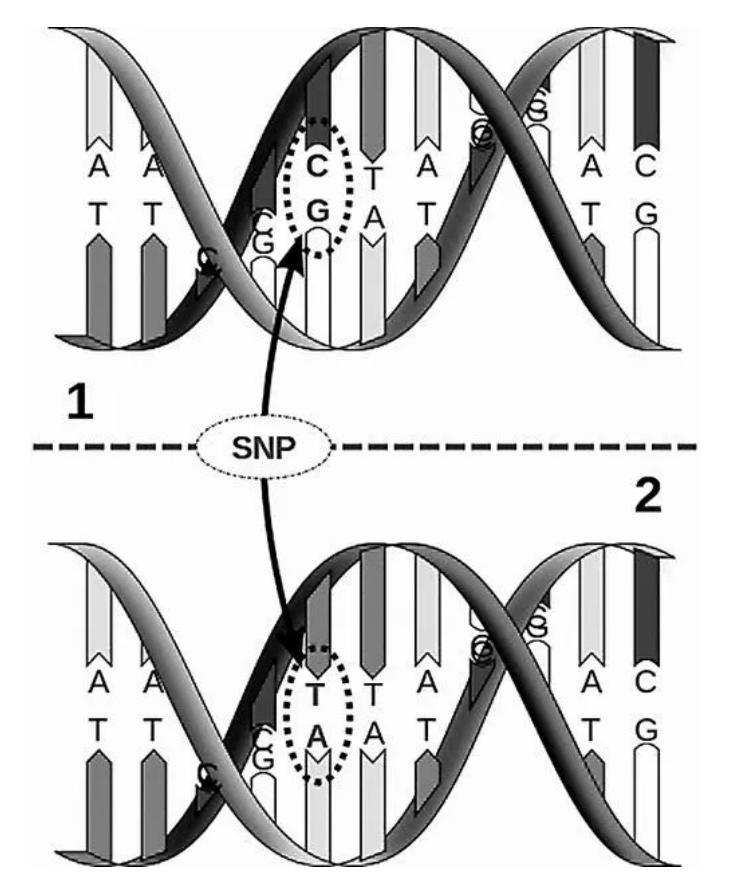
SNP (polimorfismo de nucleótido único), que podemos ver polo seu nome completo en inglés, refírese á variación ou polimorfismo dun único nucleótido.Tamén ten un nome separado, chamado SNV (variación de nucleótido único).Nalgúns estudos en humanos, só os que teñen unha frecuencia poboacional superior ao 1% reciben o nome de SNP, pero en liñas xerais pódense mesturar ambos.Polo tanto, podemos dicir que SNP, polimorfismo de nucleótido único, refírese á mutación na que un nucleótido do xenoma é substituído por outro nucleótido.Por exemplo, na figura seguinte, un par de bases AT substitúese por un par de bases GC, que é un sitio SNP.
Imaxe
Non obstante, se se trata dun "polimorfismo dun só nucleótido" ou dunha "variación dun só nucleótido", é relativamente falando, polo que os datos do SNP necesitan a resecuenciación do xenoma como base, é dicir, os datos de secuenciación son secuenciados despois de que o xenoma do individuo sexa secuenciado.En comparación co xenoma, o sitio que difire do xenoma detéctase como un sitio SNP.
En termos de SNP en vexetais,o kit Plant Direct PCRpódese usar para unha detección rápida.
En canto aos tipos de mutación, o SNP inclúe a transición e a transversión.A transición refírese á substitución de purinas por purinas ou pirimidinas por pirimidinas.A transversión refírese á substitución mutua entre purinas e pirimidinas.A frecuencia de aparición será diferente e a probabilidade de transición será maior que a de transversión.
En canto a onde se produce o SNP, os diferentes SNP terán diferentes efectos sobre o xenoma.Os SNP que se producen na rexión interxénica, é dicir, a rexión entre xenes do xenoma, poden non afectar a función do xenoma, e as mutacións no intrón ou na rexión promotora augas arriba do xene poden ter un certo impacto sobre o xene;As mutacións que se producen nas rexións exóns dos xenes, dependendo de se provocan cambios nos aminoácidos codificados, teñen diferentes efectos sobre as funcións dos xenes.(Por suposto, aínda que dous SNP causan diferenzas nos aminoácidos, teñen diferentes efectos sobre a estrutura da proteína e, finalmente, os efectos sobre o fenotipo biolóxico poden ser moi diferentes).
Non obstante, o número de SNP que se producen nas localizacións xenéticas adoita ser significativamente menor que o de localizacións non xenéticas, porque un SNP que afecta a función dun xene adoita ter un impacto negativo na supervivencia do individuo, polo que o individuo portador deste SNP no grupo Entre eles foi eliminado.
Por suposto, para os organismos diploides, os cromosomas existen en pares, pero é imposible que un par de cromosomas sexa exactamente o mesmo para todas as bases.Polo tanto, algúns SNP tamén aparecerán heterocigotos, é dicir, hai dúas bases nesta posición do cromosoma.Nun grupo, os xenotipos de SNP de diferentes individuos agréganse, o que se converte na base para a maioría das análises posteriores.En combinación con trazos, pódese xulgar se o SNP como marcador molecular está ligado a trazos, se pode xulgar o QTL (locus cuantitativo do trazo) do trazo e se pode realizar GWAS (estudo de asociación de todo o xenoma) ou a construción de mapas xenéticos;O SNP pode usarse como marcador molecular Xulgar a relación evolutiva entre individuos;pode cribar SNP funcionais e estudar mutacións relacionadas coa enfermidade;pode usar os cambios de frecuencia dos alelos SNP ou as taxas heterocigotas e outros indicadores para determinar as rexións seleccionadas no xenoma... etc., combinados co presente Co desenvolvemento da secuenciación de alto rendemento, pódense obter centos de miles ou máis sitios SNP a partir dun conxunto de datos de secuenciación.Pódese dicir que o SNP converteuse agora na pedra angular da investigación xenética das poboacións.
Por suposto, os cambios nas bases do xenoma non sempre son a substitución dunha base por outra (aínda que esta é a máis común).Tamén é posible que falten unha ou algunhas bases, ou dúas bases.Varias outras bases foron inseridas no medio.Este pequeno rango de insercións e delecións chámase colectivamente InDel (inserción e eliminación), que se refire especificamente á inserción e eliminación de fragmentos curtos (unha ou varias bases).InDel que ocorre na localización dun xene tamén pode ter un impacto na función do xene, polo que ás veces InDel tamén pode desempeñar un papel importante na investigación.Pero, en xeral, o estado do SNP como pedra angular da xenética de poboacións aínda é inquebrantable.
Hora de publicación: 27-Ago-2021