1. Coñecementos básicos (se queres ver a parte experimental, transfire directamente á segunda parte)
Como reacción derivada da PCR convencional, a PCR en tempo real monitoriza principalmente o cambio da cantidade de produto de amplificación en cada ciclo da reacción de amplificación da PCR en tempo real a través do cambio do sinal de fluorescencia e analiza cuantitativamente o modelo inicial a través da relación entre o valor ct e a curva estándar.
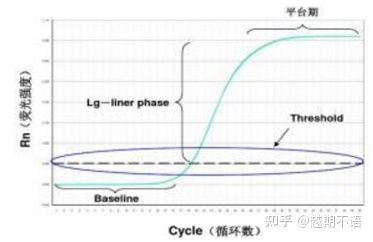
Os datos específicos da RT-PCR sonliña de base, umbral de fluorescenciaeValor Ct.
| liña base: | O valor de fluorescencia do ciclo 3-15 é a liña base (línea de base), que é causada polo erro ocasional da medición. |
| Limiar (limiar): | Refírese ao límite de detección de fluorescencia establecido nunha posición adecuada na rexión de crecemento exponencial da curva de amplificación, xeralmente 10 veces a desviación estándar da liña base. |
| Valor CT: | É o número de ciclos de PCR cando o valor de fluorescencia en cada tubo de reacción alcanza o limiar. O valor Ct é inversamente proporcional á cantidade de modelo inicial. |
Métodos de etiquetaxe comúns para RT-PCR:
| método | vantaxe | deficiencia | ámbito de aplicación |
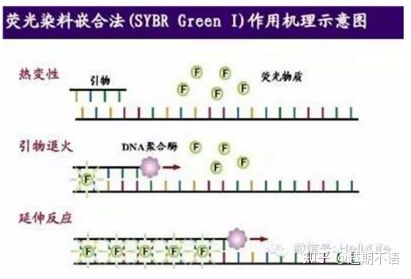
| SYBR VerdeⅠ | Ampla aplicabilidade, sensible, barato e cómodo | Os requisitos de imprimación son altos, propensos a bandas non específicas | É axeitado para a análise cuantitativa de varios xenes diana, a investigación sobre a expresión xénica e a investigación en animais e plantas recombinantes transxénicos. |
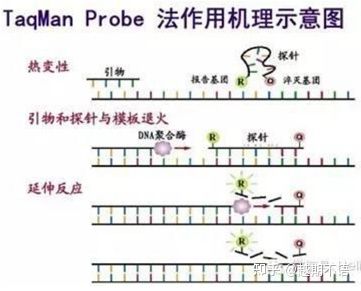
| TaqMan | Boa especificidade e alta repetibilidade | O prezo é alto e só apto para obxectivos específicos. | Detección de patóxenos, investigación de xenes de resistencia a medicamentos, avaliación da eficacia dos fármacos, diagnóstico de enfermidades xenéticas. |
| faro molecular | Alta especificidade, fluorescencia, fondo baixo | O prezo é alto, só é adecuado para un propósito específico, o deseño é difícil e o prezo é alto. | Análise de xenes específicos, análise de SNP |
2. Pasos experimentais
2.1 Sobre a agrupación experimental- Debe haber varios pozos no grupo, e debe haber repeticións biolóxicas.
| ① | Control en branco | Úsase para detectar o estado de crecemento celular en experimentos |
| ② | ARNsi de control negativo (secuencia de ARNsi non específica) | Demostrar a especificidade da acción do ARNi.O siRNA pode inducir unha resposta de estrés non específica a unha concentración de 200 nM. |
| ③ | Control de reactivos de transfección | Excluír a toxicidade do reactivo de transfección para as células ou o efecto sobre a expresión do xene diana |
| ④ | siRNA contra o xene diana | Derrubar a expresión do xene diana |
| ⑤ (opcional) | siRNA positivo | Úsase para solucionar problemas de funcionamento e sistemas experimentais |
| ⑥ (opcional) | siRNA de control fluorescente | A eficacia da transfección celular pódese observar cun microscopio |
2.2 Principios de deseño de imprimacións
| Tamaño do fragmento amplificado | Preferiblemente a 100-150 pb |
| Lonxitude de imprimación | 18-25 pb |
| Contido GC | 30%-70%, preferentemente 45%-55% |
| Valor Tm | 58-60 ℃ |
| Secuencia | Evite T/C continuo;A/G continuo |
| 3ª secuencia final | Evite rico en GC ou rico en AT;a base terminal é preferentemente G ou C;o mellor é evitar T |
| Complementariedade | Evite secuencias complementarias de máis de 3 bases dentro do cebador ou entre dous cebadores |
| Especificidade | Use a busca explosiva para confirmar a especificidade do cebador |
①SiRNA é específico da especie, e as secuencias das diferentes especies serán diferentes.
②SiRNA está envasado en po liofilizado, que se pode almacenar de forma estable durante 2-4 semanas a temperatura ambiente.
2.3 Ferramentas ou reactivos que cómpre preparar previamente
| Primer (referencia interna) | Incluíndo adiante e atrás dous |
| Cebadores (xene obxectivo) | Incluíndo adiante e atrás dous |
| Target Si RNA (3 tiras) | Xeralmente, a empresa sintetizará 3 tiras e despois escollerá unha das tres mediante RT-PCR |
| Kit de Transfección | Lipo2000 etc. |
| Kit de extracción rápida de ARN | Para extracción de ARN despois da transfección |
| Kit de transcrición inversa rápida | para a síntese de cDNA |
| Kit de amplificación por PCR | 2×Super SYBR Green qPCR Master Mix |
2.4 En canto ás cuestións ás que se debe prestar atención nas etapas experimentais específicas:
①Proceso de transfección de ARNsi
1. Para o enchapado, pode escoller unha placa de 24 pocillos, unha placa de 12 pocillos ou unha placa de 6 pocillos (a concentración media de ARN proposta en cada pocillo dunha placa de 24 pocillos é de aproximadamente 100-300 ng/uL), e a densidade de transfección óptima das células é de ata un 60% -80% aproximadamente.
2. Os pasos de transfección e os requisitos específicos están estrictamente de acordo coas instrucións.
3. Despois da transfección, pódense recoller mostras en 24-72 horas para a detección de ARNm (RT-PCR) ou a detección de proteínas en 48-96 horas (WB)
② Proceso de extracción de ARN
1. Evitar a contaminación por encimas esóxenos.Inclúe principalmente o uso de máscaras e luvas estrictamente;utilizando puntas de pipeta esterilizadas e tubos EP;a auga utilizada no experimento debe estar libre de RNase.
2. Recoméndase facer dúas veces como se suxire no kit de extracción rápida, o que realmente mellorará a pureza e o rendemento.
3. O líquido residual non debe tocar a columna de ARN.
③ Cuantificación de ARN
Despois de extraer o ARN, pódese cuantificar directamente con Nanodrop e a lectura mínima pode ser de tan só 10 ng/ul.
④Proceso de transcrición inversa
1. Debido á alta sensibilidade da RT-qPCR, débense facer polo menos 3 pozos paralelos para cada mostra para evitar que o Ct posterior sexa demasiado diferente ou que o SD sexa demasiado grande para a análise estatística.
2. Non conxelar e desconxelar Master Mix repetidamente.
3. Cada tubo/orificio debe ser substituído por unha nova punta!Non use continuamente a mesma punta de pipeta para engadir mostras.
4. A película unida á placa de 96 pozos despois de engadir a mostra debe suavizarse cunha placa.O mellor é centrifugar antes de poñelo na máquina, para que o líquido da parede do tubo poida baixar e eliminar as burbullas de aire.
⑤Análise de curva común
| Sen período de crecemento logarítmico | Posiblemente alta concentración de modelo |
| Sen valor CT | Pasos incorrectos para detectar sinais fluorescentes; degradación de cebadores ou sondas: a súa integridade pódese detectar mediante electroforese PAGE; cantidade insuficiente de modelo; degradación de modelos: evitando a introdución de impurezas e a conxelación e desconxelación repetidas na preparación da mostra; |
| Ct>38 | Baixa eficiencia de amplificación;O produto da PCR é demasiado longo;varios compoñentes da reacción son degradados |
| Curva de amplificación lineal | As sondas poden degradarse parcialmente por ciclos repetidos de conxelación-descongelación ou exposición prolongada á luz |
| A diferenza de buratos duplicados é particularmente grande | A solución de reacción non está completamente derretida ou a solución de reacción non se mestura;o baño termal do instrumento de PCR está contaminado por substancias fluorescentes |
2.5 Sobre a análise de datos
A análise de datos da qPCR pódese dividir en cuantificación relativa e cuantificación absoluta.Por exemplo, as células do grupo de tratamento en comparación coas células do grupo de control,
Cantas veces cambia o ARNm do xene X, esta é a cuantificación relativa;nun determinado número de células, o ARNm do xene X
Cantos exemplares hai, isto é unha cuantificación absoluta.Normalmente o que máis usamos no laboratorio é o método cuantitativo relativo.Normalmente,o método 2-ΔΔctúsase máis nos experimentos, polo que só se presentará este método en detalle aquí.
Método 2-ΔΔct: O resultado obtido é a diferenza na expresión do xene diana no grupo experimental en relación co xene diana no grupo control.Requírese que as eficiencias de amplificación tanto do xene obxectivo como do xene de referencia interno sexan próximas ao 100% e a desviación relativa non debe exceder o 5%.
O método de cálculo é o seguinte:
Δct grupo control = valor ct do xene diana no grupo control – valor ct do xene de referencia interno no grupo control
Δct grupo experimental = valor ct do xene diana no grupo experimental – valor ct do xene de referencia interno no grupo experimental
ΔΔct=Δct grupo experimental-Δct grupo control
Finalmente, calcula o múltiplo da diferenza no nivel de expresión:
Cambiar Fold=2-ΔΔct (correspondente á función de Excel é POTENCIA)
Produtos relacionados:
Hora de publicación: 20-mai-2023