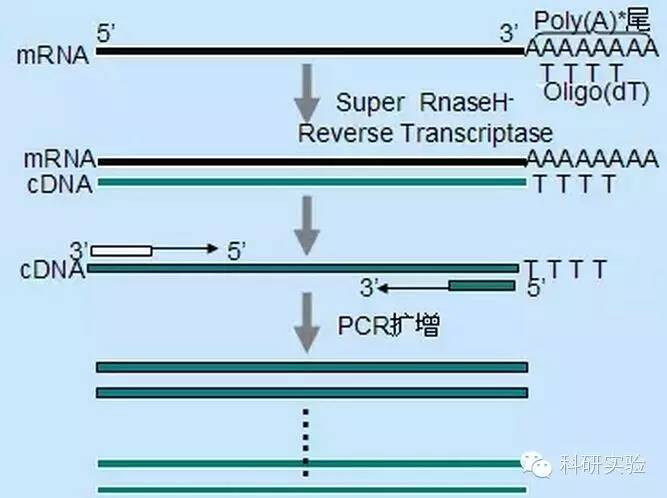
Ⅰ. Aumenta a sensibilidade do sistema de reacción:
1. Separar ARN de alta calidade:
A síntese exitosa de ADNc provén de ARN de alta calidade.O ARN de alta calidade debe garantir polo menos un total máis longo e non contén inhibidores que non conteñan encimas de rexistro, como EDTA ou SDS.A calidade do ARN determina o valor máximo da información da secuencia que podes transcribir ao cDNA.O método xeral de purificación de ARN é un método escalonado de uso de isoocianato/acidofenol.Para evitar a contaminación da RNase, o ARN separado dunha mostra rica en RNase (como o páncreas) require almacenamento de formaldehido para gardar ARN de alta calidade, o que é aínda máis para o almacenamento a longo prazo.O ARN extraído do fígado de rato degradouse basicamente despois dunha semana de almacenamento en auga, mentres que o ARN extraído do bazo da rata permaneceu estable despois de tres anos de almacenamento na auga.Ademais, as transcricións de máis de 4 kb son máis sensibles á degradación da RNase traza que as pequenas transcricións.Para aumentar a estabilidade da mostra de ARN de almacenamento, o ARN pódese disolver nunha metamamina de ión, e almacénase a -70 °C.O tilido usado para gardar ARN non debe conter un obxecto diverso que degrade o ARN.O ARN, que se deriva do páncreas, pódese gardar en metalmamina durante polo menos un ano.Cando estea listo para usar o ARN, pode usar os seguintes métodos para precipitar o ARN: engadir NaCl a 0,2 m e 4 veces o volume de etanol, coloque a temperatura ambiente durante 3-5 minutos e centrífuga a 10.000 × g durante 5 minutos.
2. Use transcritase inversa sen actividade RNaseH (RNaseH-):
Os inhibidores da RNase adoitan engadirse ás reaccións de transcrición inversa para aumentar a lonxitude e o rendemento da síntese de ADNc.O inhibidor da RNase engádese na primeira reacción de síntese en cadea en presenza de tampóns e axentes reductores como o DTT porque o proceso de síntese de pre-ADNc desnaturaliza o inhibidor, liberando así as RNases unidas que degradan o ARN.O inhibidor da proteína RNase só impide a degradación do ARN pola RNase A, B, C, e non prevén as RNase na pel, polo que hai que ter coidado de non introducir RNases polos dedos a pesar do uso destes inhibidores.
A transcriptase inversa cataliza a conversión de ARN en ADNc.Tanto o M-MLV como o AMV teñen actividade RNaseH endóxena ademais da súa propia actividade polimerase.A actividade da RNaseH compite coa actividade da polimerase polas cadeas heterocigotas formadas entre os modelos de ARN e os cebadores de ADN ou as cadeas de extensión de ADNc, e degrada as cadeas de ARN: ARN nos complexos de ADN.Os moldes de ARN degradados pola actividade da RNaseH xa non poden usarse como substratos eficaces para a síntese de ADNc, o que reduce o rendemento e a duración da síntese de ADNc.Así, eliminar ou reducir moito a actividade da RNaseH da transcriptase inversa sería de gran beneficio.
A transcriptase inversa SuperScriptⅡ, a transcriptase inversa MMLV de RNaseH- e a transcriptase inversa thermoScript, AMV de RNaseH- produciron máis ADNc de lonxitude total que MMLV e AMV.A sensibilidade da RT-PCR vese afectada pola cantidade de ADNc sintetizado.ThermoScript é moito máis sensible que AMV.O tamaño dos produtos de RT-PCR está limitado pola capacidade da transcriptase inversa para sintetizar ADNc, especialmente cando se clonan Cdnas máis grandes.En comparación co MMLV, SuperScripⅡ aumentou significativamente o rendemento dos produtos longos de RT-PCR.A transcriptase inversa da RNaseH- tamén aumenta a estabilidade térmica, polo que a reacción pode levarse a cabo a temperaturas superiores ao normal de 37-42 ℃.Baixo as condicións de síntese suxeridas, utilizáronse cebadores oligo(dT) e 10μCi [alfa-p]dCTP.A produción total da primeira cadea calculouse mediante o método de precipitación TCA.Analizouse o ADNc de lonxitude completa mediante a eliminación de tiras clasificadas por tamaño e contando nun xel de agarosa alcalina.
3. Aumenta a temperatura de conservación da calor da transcrición inversa:
A temperatura de retención máis alta axuda a abrir a estrutura secundaria do ARN e aumenta o rendemento da reacción.Para a maioría dos modelos de ARN, manter o ARN e o cebador a 65 °C sen tampón nin sal e despois arrefrialos rapidamente en xeo elimina a maioría das estruturas secundarias e permite que os cebadores se unan.Non obstante, algúns modelos aínda teñen estrutura secundaria, mesmo despois da desnaturalización térmica.A amplificación destes modelos difíciles pódese realizar mediante a transcriptase inversa ThermoScript e colocando a reacción da transcriptase inversa a temperaturas máis altas para mellorar a amplificación.As temperaturas de mantemento máis altas tamén poden aumentar a especificidade, especialmente cando a síntese de ADNc se realiza mediante cebadores específicos de xenes (GSPS) (ver capítulo 3).Se utiliza GSP, asegúrese de que o valor de Tm do cebador é o mesmo que a temperatura de mantemento esperada.Non use oligo(dT) e cebadores aleatorios por riba de 60 ℃.Os cebadores aleatorios deben manterse a 25 ℃ durante 10 minutos antes de aumentar a 60 ℃.Ademais de usar temperaturas de transcrición inversa máis altas, pódese mellorar a especificidade transferindo directamente a mestura de ARN/cebador desde a temperatura de desnaturalización de 65 ℃ á temperatura de mantemento da transcrición inversa e engadindo unha mestura de reacción 2x prequentada (síntese de iniciación térmica de ADNc).Este enfoque axuda a evitar o emparellamento de bases intermoleculares que se produce a temperaturas máis baixas.O uso dun instrumento de PCR simplifica os moitos interruptores de temperatura necesarios para a RT-PCR.
A polimerase Tth estabilizada por calor actúa como ADN polimerase en presenza de Mg2+ e ARN polimerase en presenza de Mn2+.Pode manter a calor ata 65 ℃.Non obstante, a presenza de Mn2+ durante a PCR reduce a fidelidade, o que fai que a polimerase Tth sexa menos apta para a amplificación de alta precisión, como a clonación de ADNc.Ademais, a Tth é menos eficiente na transcrición inversa, o que reduce a sensibilidade, e dado que un único encima pode realizar a transcrición inversa e a PCR, as reaccións de control sen transcrición inversa non se poden utilizar para distinguir os produtos amplificados do ADNc dos do ADN xenómico contaminado.
4. Aditivo que promove a transcrición inversa:
A adición de aditivos, incluíndo glicerina e DMSO, á primeira reacción de síntese en cadea pode reducir a estabilidade da dobre cadea do ácido nucleico e desenrolar a estrutura secundaria do ARN.Pódese engadir ata un 20% de glicerina ou un 10% de DMSO sen afectar a actividade de SuperScriptⅡ ou MMLV.O AMV tamén pode tolerar ata un 20% de glicerol sen reducir a actividade.Para maximizar a sensibilidade da RT-PCR na reacción de transcrición inversa SuperScriptⅡ, pódese engadir un 10% de glicerol e illar a 45 ℃.Se se engade 1/10 do produto da reacción de retrotranscrición á PCR, a concentración de glicerol na reacción de amplificación é do 0,4%, o que non é suficiente para inhibir a PCR.
5. Procesamento de RNaseH:
A sensibilidade pódese mellorar tratando as reaccións de síntese de ADNc con RNaseH antes da PCR.Para algúns modelos, pénsase que o ARN na reacción de síntese de ADNc impide a unión de produtos amplificados, nese caso o tratamento con RNaseH pode aumentar a sensibilidade.Xeralmente, o tratamento con RNaseH é necesario para a amplificación dunha plantilla diana de ADNc de lonxitude completa relativamente longa, como a esquerose tuberosaⅡ con copia baixa.Para este modelo difícil, RNaseH mellorou o sinal xerado polo cDNA sintetizado por SuperScriptⅡ ou AMV.Para a maioría das reaccións de RT-PCR, o tratamento con RNaseH é opcional porque a etapa de desnaturalización da PCR illada a 95 ℃ normalmente hidroliza o ARN do complexo ARN: ADN.
6. Métodos mellorados para detectar pequenas cantidades de ARN:
A RT-PCR é particularmente difícil cando só se dispón de pequenas cantidades de ARN.A adición de glicóxeno como vehículo durante a separación do ARN axuda a aumentar o rendemento de pequenas mostras.Pódese engadir un glicóxeno sen RNase ao mesmo tempo que Trizol.O glicóxeno é soluble en auga e pode permanecer na fase acuosa con ARN para axudar na precipitación posterior.A concentración recomendada de glicóxeno libre de RNase é de 250 μg/ml para mostras de menos de 50 mg de tecido ou 106 células cultivadas.
A adición de BSA acetilado ás reaccións de transcrición inversa usando SuperScriptⅡ pode aumentar a sensibilidade, e para pequenas cantidades de ARN, reducir a cantidade de SuperScriptⅡ e engadir 40 unidades de inhibidor de nuclease RnaseOut pode mellorar o nivel de detección.Se se usa glicóxeno na separación do ARN, aínda se recomenda a adición de inhibidores de BSA ou RNase para revertir as reaccións de transcrición usando SuperScriptⅡ.
Ⅱ. Aumentar a especificidade da RT-PCR
1. Síntese cNDA:
Pódense usar tres métodos diferentes para iniciar a síntese de ADNc da primeira cadea, e a especificidade relativa de cada método afecta á cantidade e tipo de ADNc sintetizado.
O método de imprimación aleatoria é o menos específico dos tres métodos.Os cebadores son recocidos en múltiples sitios ao longo do transcrito para producir ADNc curto e de lonxitude parcial.Este método utilízase a miúdo para obter secuencias terminais 5′ e ADNc de moldes de ARN con rexións estruturais secundarias ou con sitios de terminación que non se poden replicar da transcriptase inversa.Para obter o ADNc máis longo, a relación entre cebadores e ARN en cada mostra de ARN debe determinarse empíricamente.A concentración inicial de cebadores aleatorios varía de 50 a 250 ng por sistema de reacción de 20 μl.Dado que o ADNc sintetizado a partir do ARN total usando cebadores aleatorios é principalmente ARN ribosómico, polo xeral se selecciona poli(A)+ARN como molde.
O inicio do oligo(dT) é máis específico que os cebadores aleatorios.Hibridízase coa cola poli(A) que se atopa no extremo 3′ do ARNm na maioría das células eucariotas.Debido a que o poli(A)+ARN é aproximadamente do 1% ao 2% do ARN total, a cantidade e complexidade do ADNc é moito menor que se se usasen cebadores aleatorios.Debido á súa alta especificidade, o oligo(dT) xeralmente non require optimización para a relación ARN-cebador e a selección de poli(A)+.Recoméndase usar 0,5 μg de oligo(dT) por 20 μl de sistema de reacción.oligo(dT)12-18 é axeitado para a maioría da RT-PCR.O sistema ThermoScript RT-PCR proporciona oligo(dT)20 debido á súa boa estabilidade térmica e é adecuado para temperaturas de mantemento máis altas.
Os cebadores específicos de xenes (GSP) son os mellores cebadores específicos para a etapa de transcrición inversa.O GSP é un oligonucleósido antisentido que pode hibridar especificamente con secuencias de destino de ARN, en lugar de recocer todos os Rnas como cebadores aleatorios ou oligo(dT).As regras utilizadas para deseñar cebadores de PCR tamén se aplican ao deseño da reacción de transcrición inversa GSP.O GSP pode ser a mesma secuencia que o cebador de amplificación recocido ao final do ARNm3', ou GSP pode ser deseñado para ser recocido augas abaixo co cebador de amplificación inversa.Para algúns obxectos amplificados, é necesario deseñar máis dun cebador antisentido para unha RT-PCR exitosa porque a estrutura secundaria do ARN diana pode impedir a unión do cebador.Suxírese usar 1 pmol de GSP antisentido no primeiro sistema de reacción de síntese en cadea de 20 μl.
2. Aumenta a temperatura de conservación da calor da transcrición inversa:
Para aproveitar ao máximo a especificidade do GSP, débese utilizar a transcriptase inversa cunha alta estabilidade térmica.A transcriptase inversa termoestable pódese illar a temperaturas máis altas para aumentar o rigor da reacción.Por exemplo, se un GSP é recocido a 55 °C, entón a especificidade do GSP non se utiliza completamente se a transcrición inversa se realiza a 37 °C con baixo rigor usando AMV ou M-MLV.Non obstante, SuperScripⅡ e ThermoScript poden reaccionar a 50 ℃ ou máis, o que elimina os produtos inespecíficos producidos a temperaturas máis baixas.Para obter a máxima especificidade, a mestura de ARN/cebador pódese transferir directamente desde a temperatura de desnaturalización de 65 ℃ á temperatura de mantemento da transcrición inversa coa adición dunha mestura de reacción prequentada 2 veces (iniciación térmica da síntese de ADNc).Isto axuda a evitar o emparellamento de bases entre moléculas a baixas temperaturas.O uso dun instrumento de PCR simplifica as moitas transicións de temperatura necesarias para a RT-PCR.
3. Reducir a contaminación do ADN xenómico:
Unha dificultade potencial coa RT-PCR é que o ARN contamina o ADN xenómico.O uso de mellores métodos de separación de ARN, como o reactivo Trizol, reduce a contaminación do ADN xenómico nas preparacións de ARN.Para evitar produtos producidos a partir de ADN xenómico, o ARN pódese tratar con DnasⅠ de grao de amplificación para eliminar o ADN contaminado antes da transcrición inversa.As mostras mantivéronse a 65 ℃ en EDTA 2,0 mM durante 10 min para terminar a dixestión da DNaseⅠ.O EDTA quela os ións magnesio para evitar a hidrólise do ARN dependente do ión magnesio que se produce a altas temperaturas.
Para separar o ADNc amplificado do produto de amplificación do ADN do xenoma, pódense deseñar cebadores que se asoman por separado co exón separado.Os produtos de PCR derivados do ADNc serán máis curtos que os derivados do ADN xenómico contaminado.Tamén se realiza un experimento controlado sen transcrición inversa en cada molde de ARN para determinar se un determinado fragmento procede de ADN xenómico ou de ADNc.Os produtos da PCR obtidos en ausencia de transcrición inversa derivan do xenoma.
Produto relacionado
-O kit dun paso permite realizar a transcrición inversa e a PCR no mesmo tubo.Só precisa engadir ARN modelo, cebadores específicos de PCR e ddH sen RNase2O.
-A análise cuantitativa de ARN en tempo real pódese realizar de forma rápida e precisa.
-O kit usa un reactivo de transcrición inversa Foregene único e Foregene HotStar Taq DNA Polymerase combinado cun sistema de reacción único para mellorar eficazmente a eficiencia e especificidade da amplificación da reacción.
-O sistema de reacción optimizado fai que a reacción teña unha maior sensibilidade de detección, unha maior estabilidade térmica e unha mellor tolerancia.
-Capacidade eficiente para eliminar gDNA, que pode eliminar gDNA do modelo en 2 minutos.
-Eficiente sistema de transcrición inversa, só leva 15 minutos completar a síntese da primeira cadea de ADNc.
-Modelos complexos: os modelos con alto contido de GC e estrutura secundaria complexa tamén se poden reverter con alta eficiencia.
-Sistema de transcrición inversa de alta sensibilidade, os modelos de nivel pg tamén poden obter ADNc de alta calidade.
-O sistema de transcrición inversa ten unha alta estabilidade térmica, a temperatura de reacción óptima é de 42 ℃ e aínda ten un bo rendemento de transcrición inversa a 50 ℃.
Hora de publicación: Mar-07-2023