Material de partida: ARN
A PCR de transcrición inversa cuantitativa (RT-qPCR) é un método experimental que se usa en experimentos de PCR usando ARN como material de partida.Neste método, o ARN total ou ARN mensaxeiro (ARNm) transcríbese primeiro a ADN complementario (ADNc) mediante a transcriptase inversa.Posteriormente, realizouse unha reacción qPCR usando o ADNc como modelo.A RT-qPCR utilizouse nunha variedade de aplicacións de bioloxía molecular, incluíndo análise de expresión xénica, validación da interferencia de ARN, validación de microarrays, detección de patóxenos, probas xenéticas e investigación de enfermidades.
Métodos dun e dous pasos para RT-qPCR
A RT-qPCR pódese realizar mediante un método dun ou dous pasos.A RT-qPCR dun paso combina a transcrición inversa e a amplificación por PCR, o que permite que a transcriptase inversa e a ADN polimerase completen a reacción no mesmo tubo nas mesmas condicións de tampón.A RT-qPCR dun paso só require o uso de cebadores específicos de secuencia.Na RT-qPCR en dous pasos, a transcrición inversa e a amplificación por PCR realízanse en dous tubos, utilizando diferentes tampóns optimizados, condicións de reacción e estratexias de deseño de cebadores.
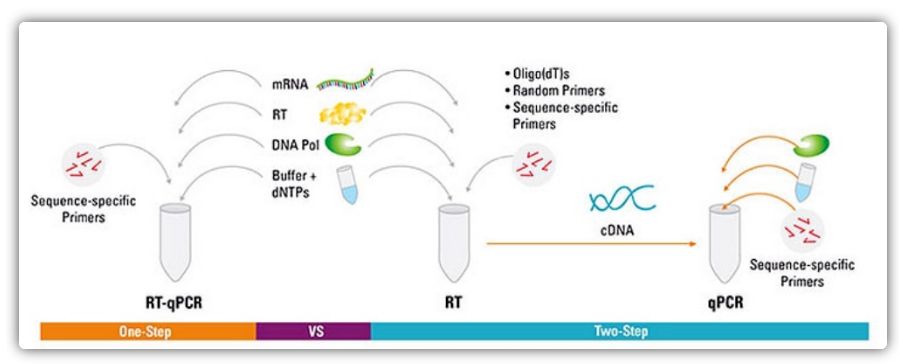
| Vantaxe | Desvantaxe | |
| Un Paso | Este método ten menos erro experimental xa que ambas reaccións fanse nun tubo
Menos pasos de pipeteo reducen o risco de contaminación
Adecuado para amplificación/screening de alto rendemento, rápido e reproducible | As reaccións en dous pasos non se poden optimizar por separado
Dado que as condicións de reacción vense comprometidas ao combinar a reacción en dúas etapas, a sensibilidade non é tan boa como a do método en dúas etapas.
O número de obxectivos detectados por unha única mostra é pequeno |
| Dous Pasos | Capacidade de crear bibliotecas de ADNc estables que se poden almacenar durante longos períodos de tempo e usarse en múltiples reaccións
Os xenes obxectivo e os xenes de referencia pódense amplificar a partir da mesma biblioteca de ADNc sen necesidade de varias bibliotecas de ADNc
Tampóns de reacción e condicións de reacción que permiten a optimización de carreiras de reacción únicas
Selección flexible das condicións de activación | O uso de varios tubos e máis pasos de pipeteo aumenta o risco de contaminación do ADN. e lento.
Require máis optimización que o método dun paso |
Produtos relacionados:
RT-qPCR Easyᵀᴹ (Un paso)-SYBR Green I
RT-qPCR Easyᵀᴹ (Un paso)-Taqman
RT Easyᵀᴹ I Master Premix para a síntese de CDNA da primeira cadea
Real Time PCR Easyᵀᴹ-SYBR Green I Kit
PCR en tempo real Easyᵀᴹ-Taqman
Selección de ARN total e ARNm
Ao deseñar un experimento de RT-qPCR, é importante decidir se se usa ARN total ou ARNm purificado como molde para a transcrición inversa.Aínda que o ARNm pode proporcionar unha sensibilidade lixeiramente maior, o ARN total aínda se usa con frecuencia.A razón disto é que o ARN total ten unha vantaxe máis importante como material de partida que o ARNm.En primeiro lugar, o proceso require menos pasos de purificación, o que garante unha mellor recuperación cuantitativa do modelo e unha mellor normalización dos resultados aos números de células de inicio.En segundo lugar, evita o paso de enriquecemento do ARNm, o que pode evitar a posibilidade de resultados sesgados debido ás diferentes recuperacións de diferentes ARNm.En xeral, dado que na maioría das aplicacións a cuantificación relativa do xene diana é máis importante que a sensibilidade absoluta da detección, o ARN total é máis axeitado na maioría dos casos.
Cebador de transcrición inversa
No método de dúas etapas, pódense usar tres métodos diferentes para cebar a reacción de ADNc: cebadores oligo(dT), cebadores aleatorios ou cebadores específicos de secuencia.Normalmente, os cebadores oligo(dT) e os cebadores aleatorios úsanse en combinación.Estes cebadores se asoman á cadea de ARNm do molde e proporcionan transcriptase inversa cun punto de partida para a síntese.
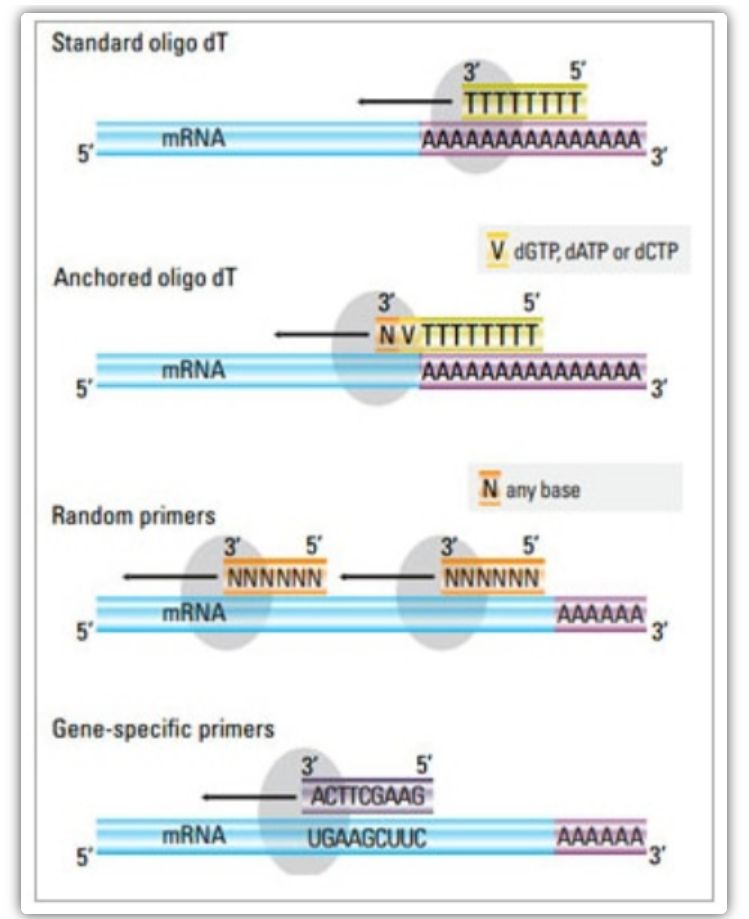
| Selección de base | Estrutura e función | Vantaxe | Desvantaxe |
| Cebador oligo(dT) (ou cebador oligo(dT) ancorado) | Recoición estendida aos residuos de timina na cola poli(A) do ARNm;O cebador oligo(dT) de áncora contén un G, C ou A no extremo 3′ (sitio de ancoraxe) | Síntese de ADNc de lonxitude total a partir de ARNm de cola poli(A).
Aplicable cando hai menos material de partida dispoñible
O sitio de ancoraxe garante que o cebador oligo(dT) se une á cola poli(A) 5′ do ARNm | Só apto para amplificar xenes con colas poli(A).
Obter ADNc truncado do sitio de cebado*2 en poli(A)
Sesgado para unirse ao extremo 3′*
*Esta posibilidade redúcese ao mínimo se se utilizan cebadores oligo(dT) ancorados |
| cebador aleatorio
| De 6 a 9 bases de lonxitude, que poden recocerse en múltiples sitios durante a transcrición do ARN | Recollido a todos os ARN (ARNt, ARNr e ARNm)
Axeitado para transcricións cunha estrutura secundaria significativa ou cando se dispón de menos material de partida
Alto rendemento de cDNA | O ADNc transcríbese inversamente a partir de todo o ARN, o que normalmente non se desexa e pode diluír o sinal do ARNm diana.
obter ADNc truncado |
| cebadores específicos de secuencia | Cebadores personalizados dirixidos a secuencias de ARNm específicas | biblioteca específica de ADNc
Mellorar a sensibilidade
Usando cebadores de qPCR inversos | Só limitado á síntese dun único xene diana |
Transcriptase inversa
A transcriptase inversa é un encima que usa ARN para sintetizar ADN.Algunhas transcriptases inversas teñen actividade RNase e poden degradar as febras de ARN nas cadeas híbridas de ARN-ADN despois da transcrición.Se non ten actividade enzimática RNase, pódese engadir RNaseH para unha maior eficiencia da qPCR.Os encimas comúnmente utilizados inclúen a transcriptase inversa do virus da leucemia murina de Moloney e a transcriptase inversa do virus do mieloblastoma aviar.Para a RT-qPCR, é ideal elixir unha transcriptase inversa con maior termoestabilidade, de xeito que a síntese de ADNc se poida realizar a temperaturas máis altas, garantindo unha transcrición exitosa de ARN con estrutura secundaria máis alta, mantendo a súa actividade total durante toda a reacción, o que resulta en maiores rendementos de ADNc.
Produtos relacionados:
Transcriptase inversa Foreasy M-MLV
Actividade da RNase H da transcriptase inversa
A RNaseH é capaz de degradar as febras de ARN dos dúplex de ARN-ADN, permitindo unha síntese eficiente de ADN de dobre cadea.Porén, cando se usa ARNm longo como molde, o ARN pode degradarse prematuramente, dando lugar a un ADNc truncado.Polo tanto, adoita ser beneficioso minimizar a actividade da RNaseH durante a clonación de ADNc se se desexa a síntese de transcritos longos.Pola contra, as transcriptases inversas con actividade RNase H adoitan ser beneficiosas para as aplicacións de qPCR porque melloran a fusión dos dúplex de ARN-ADN durante o primeiro ciclo de PCR.
Deseño de imprimación
Os cebadores de PCR utilizados para o paso qPCR na RT-qPCR deberían deseñarse idealmente para abarcar unha unión exón-exón, onde un cebador de amplificación podería abarcar potencialmente un límite exón-intrón real.Dado que as secuencias de ADN xenómico que conteñen intróns non se amplifican, este deseño reduce o risco de que se amplifican falsos positivos por contaminar o ADN xenómico.
Se non se poden deseñar cebadores para separar exóns ou límites exón-exón, pode ser necesario tratar as mostras de ARN con DNase I ou dsDNase sen RNase para eliminar a contaminación do ADN xenómico.
Control RT-qPCR
Debe incluírse un control negativo de transcrición inversa (control -RT) en todos os experimentos de RT-qPCR para detectar a contaminación do ADN (como o ADN xenómico ou os produtos de PCR de reaccións anteriores).Este control contén todos os compoñentes da reacción excepto a transcriptase inversa.Dado que a transcrición inversa non se produce con este control, se se observa amplificación por PCR, o máis probable é a contaminación do ADN.
Hora de publicación: 02-ago-2022








